雷公藤TwKS與TwCPS3在制備貝殼杉烷型二萜化合物中的應用的制作方法
本發明通過構建twcps3和twks真核及原核表達載體,通過體外酶促和酵母發酵的方法成功合成了16α-貝殼杉烷,其與赤霉素的生物合成相關。涉及雷公藤生長發育調控和活性成分的合成,屬于藥用植物基因工程領域。
背景技術:
藥用植物雷公藤(tripterygiumwilfordii.hook.f.)是一味中草藥,被廣泛用于類風濕性關節炎和炎癥的治療。萜類成分為雷公藤的主要活性成分,包括雷公藤甲素(triptolide)、雷酚內酯(triptophenolide)和雷公藤紅素(celastrol)等。現有研究表明貝殼杉烷型二萜是萜類天然產物家族中的主要組成部分,這類分子通常表現出抗菌、抗腫瘤、抗瘧、抗病毒等多種重要生物活性。從中藥活性成分開發新藥是一種很有潛力的方式,然而由于植物的生長緩慢,再加上這些有效成分在植物體中的含量不多,因而大大限制了它的發展。通過探尋和闡釋萜類成分,特別是探尋貝殼杉烷型二萜在雷公藤中的生物合成途徑及其調控機制,能夠為利用生物技術提高目標成分含量或直接生產有效成分或中間體帶來廣闊的應用空間。
赤霉素是一種四環雙萜化合物,在植物的生長調節方面具有非常重要作用的物質。現有研究表明赤霉素在植物的種子萌發,莖的生長,開花和結果具有重要的調節作用。貝殼杉烷型二萜是一類雙萜化合物,其具體種類和含量對于下游赤霉素的生物合成具有重要影響。從貝殼杉烷型二萜化合物前體到赤霉素生物合成途徑的解析有助于調控雷公藤中雷公藤甲素、雷公藤紅素等活性成分的生物合成。
技術實現要素:
本發明通過對雷公藤柯巴基焦磷酸合酶基因twcps3和雷公藤貝殼杉烯合酶基因twks進行表達載體的構建,成功合成了赤霉素的前體物16α-貝殼杉烷,這對雷公藤甲素等活性成分的生物合成調控具有重要的意義。
具體而言本發明將克隆得到的twcps3和twks基因的cdna克隆到原核表達載體pmal-c2x,構建帶有twcps3和twks基因的重組表達載體;轉入e.coli表達宿主菌,通過誘導后,表達產物經初步純化后,加入到以ggpp為底物的體外酶促反應體系中,正己烷萃取反應產物,采用gc-ms檢測到貝殼杉烷型二萜化合物的生成,經過對gc-ms圖譜的分析結果表明,twcps3和twks聯合使用能夠將ggpp轉化為貝殼杉烷型二萜類化合物,出乎意料的是,所述貝殼杉烷型二萜類化合物中除了目前已知的赤霉素前體對映貝殼杉烯以外,還能夠產生ent-16α貝殼杉烷,另外產物中還發現一定量的淚杉醇。
通過將twcps3和twks基因的orf克隆到真核表達載體pesc-trp,構建帶有twcps3和/或twks基因的重組表達載體并通過定點突變等方法,通過酵母by-t20發酵生成產物,利用氣相色譜-質譜聯用技術,分析twcps3和twks基因的功能。研究結果表明,真核表達的twcps3和twks能夠將ggpp轉化為貝殼杉烷型二萜,并且ent-16α貝殼杉烷的產量更高。本發明對雷公藤的生長發育以及活性成分的生物合成調控具有重要意義。
本發明提供一種雷公藤柯巴基焦磷酸合酶twcps3和雷公藤貝殼杉烯合酶twks在制備貝殼杉烷型二萜中的應用,其特征在于以牻牛兒基焦磷酸ggpp為底物,催化產生貝殼杉烷型二萜化合物。
其中,所述的雷公藤柯巴基焦磷酸合酶twcps3的序列為seqidno:1,雷公藤貝殼杉烯合酶twks的序列為seqidno:3。
本發明還提供雷公藤柯巴基焦磷酸合酶基因twcps3和雷公藤貝殼杉烯合酶基因twks在制備貝殼杉烷型二萜中的應用,其特征在于重組表達twcps3和twks基因獲得twcps3和twks用于制備貝殼杉烷型二萜,其中雷公藤柯巴基焦磷酸合酶基因twcps3的序列為seqidno:2,雷公藤貝殼杉烯合酶基因twks的序列為seqidno:4。
本發明所述雷公藤柯巴基焦磷酸合酶基因twcps3和雷公藤貝殼杉烯合酶基因twks在制備貝殼杉烷型二萜中的應用其中所述twcps3和twks基因通過原核表達或真核表達獲得twcps3和twks,優選將所述基因進行真核表達。
本發明所述雷公藤柯巴基焦磷酸合酶基因twcps3和雷公藤貝殼杉烯合酶基因twks在制備貝殼杉烷型二萜中的應用,利用原核表達載體pmal-c2x分別構建帶有twcps3或twks基因的重組原核表達載體;利用真核表達載體pesc-trp分別構建帶有twcps3或twks基因的重組表達載體。
本發明所述雷公藤柯巴基焦磷酸合酶twcps3/或其基因twcps3和雷公藤貝殼杉烯合酶twks/或其基因twks在制備貝殼杉烷型二萜中的應用,其中所述貝殼杉烷型二萜包括ent-貝殼杉烯和ent-16α貝殼杉烷。
本發明所述雷公藤柯巴基焦磷酸合酶twcps3和雷公藤貝殼杉烯合酶twks在制備貝殼杉烷型二萜中的應用,其特征在于產物中還包括淚杉醇。
本發明所述雷公藤柯巴基焦磷酸合酶twcps3/或其基因twcps3和雷公藤貝殼杉烯合酶twks/或其基因twks在制備貝殼杉烷型二萜中的應用,其特征在于雷公藤柯巴基焦磷酸合酶twcps3/或其基因twcps3,與雷公藤貝殼杉烯合酶twks/或其基因twks同時使用或先后使用。
本發明還提供一種酶催化制備貝殼杉烷型二萜的方法,其包括:(1)以牻牛兒基焦磷酸ggpp為底物加入雷公藤柯巴基焦磷酸合酶twcps3,正己烷萃取3次,棄有機相、取水相,氮氣吹干后進行脫磷,(2)向步驟(1)中脫磷后獲得的產物加入雷公藤貝殼杉烯合酶twks進行反應,正己烷萃取3次,取正己烷相用氮氣吹干,(3)對步驟(2)獲得的產物進行gc-ms分析。
本發明還提供一種酶催化制備貝殼杉烷型二萜的方法,其包括:(1)將twcps3基因表達盒和twks基因表達盒構建到同一個真核表達載體pesc-trp上,構建獲得重組質粒pesc-(twcps3+twks),轉化酵母篩選獲得重組酵母菌;(2)將pcr鑒定陽性的重組酵母菌進行誘導培養,收集菌液加入等體積正己烷,超聲破菌、萃取、硫酸鈉干燥、旋蒸濃縮至近干,過濾后4℃保存備用;(3)對步驟(2)獲得的產物進行gc-ms分析。
本發明所述酶催化制備貝殼杉烷型二萜的方法,其特征在于gc-ms分析的步驟中第12.99分的峰為淚杉醇,第13.03分的峰為ent-貝殼杉烯,第13.96分的峰為ent-16α-貝殼杉烷。
附圖說明
圖1茉莉酸甲酯mj誘導雷公藤二萜合酶基因不同時間的相對表達量
圖2宿主菌e.colitransb(de3)誘導表達產物sds-page蛋白電泳分析(c:陰性對照即空表達載體pmal-c2x表達產物;m:蛋白分子量標準,條帶由上往下分別為200、116、97.2、66.4、29.0、20.1、14.3、6.5kda;箭頭表示目的重組蛋白;1:重組質粒pmaltwcps1的表達;2:重組質粒pmaltwcps3的表達產物;3:重組質粒pmaltwks的表達產物;4:重組質粒pmaltwcps2的表達產物;5:重組質粒pmaltwges2表達產物;6:重組質粒pmaltwges1表達產物)
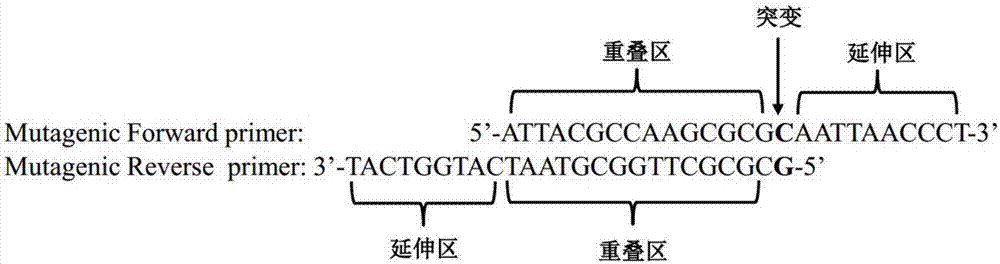
圖3定點突變引物設計
圖4雷公藤二萜合酶真核表達載體構建
圖5cps催化底物ggpp形成產物提取離子色譜圖,第13.83分的峰為cpp,質譜荷質此為275m/z。由上而下依次為twcps1、twcps2、twcps3、smcps1、atcps、空載體。
圖6(cps+ks)催化ggpp形成產物提取離子色譜圖,第13.06分的峰為次丹參酮二烯,第13.03分的峰為對映貝殼杉烯第13.83分的峰為cpp,第13.96分的峰為ent-16α-貝殼杉烷,第13.46分的峰為ggoh;質譜荷質此為272m/z。
圖7twcps2c311d、twkst318d催化ggpp形成產物提取離子色譜圖,第13.46分的峰為ggoh;質譜荷質此為275m/z、272m/z。
圖8by-t20/pesc-trp-twcps3+twks、by-t20/pesc-trp-twcps3+twksa608m、by-t20/pesc-trp發酵產物提取離子色譜圖,第13.03分的峰為對映貝殼杉烯;質譜荷質此為272m/z。
圖9by-t20/pesc-trp、by-t20/pesc-trp-twcps1、by-t20/pesc-trp-twcps3+twks、by-t20/pesc-trp-twcps3+twksa608m發酵產物提取離子色譜圖第12.99分的峰為淚杉醇,第13.03分的峰為對映貝殼杉烯,第13.83分的峰為cpp,第13.96分的峰為ent-16α-貝殼杉烷;質譜荷質此為272m/z。
具體實施方式
以下結合具體實施例,進一步闡述本發明。這些實施例僅用于說明本發明而不用于限制本發明的范圍。下列實施例中未注明具體條件的實驗方法,通常按照常規條件或按照制造廠商所建議的條件。除非另行定義,文中所使用的所有專業與科學用語與本領域熟練人員所熟悉的意義相同。此外,任何與所記載內容相似或均等的方法及材料皆可應用于本發明方法中。文中所述的較佳實施方法與材料僅作示范之用。
下述實施例中所使用的實驗方法如無特殊說明,均為常規方法。所用的材料、試劑等,如無特殊說明,均可從商業途徑得到。
雷公藤(tripterygiumwilfordiihook.f.)懸浮細胞在文獻“雷公藤4-(5’-二磷酸胞苷)-2-c-甲基-d-赤蘚醇激酶基因的全長克隆與表達分析.中國中藥雜志,2015,40(21):4165-4170”中公開過,公眾可從首都醫科大學分子生藥與中藥資源實驗室獲得。
smartertmracecdnaamplificationkit,primestargxldnapolymerase購自takara公司;peasy-bluntsimplecloningkit購自北京全式金生物技術有限公司,ggpp購自sigma公司,產品目錄號為g6025。
實施例一、雷公藤twcps3和twks原核表達載體構建(圖1)
分別以含有雷公藤twcps3和twks基因全長cdna的載體pmd-19-tps質粒為模板,用含酶切位點引物,進行pcr擴增基因編碼區(引物序列見表1),插入原核表達載體中。dna聚合酶采用高保真dna聚合酶(primestarhsdnapolymerase)。pcr參數為98℃3min,1循環;98℃10s,60℃10s,72℃2min30s,30循環;72℃7min;4℃維持。擴增產物經genejetgelextractionkit膠回收(方法如下)。
genejetgelextractionkit膠回收步驟:
(1)取pcr產物與6×loadingbuffer預混合,在1.5%瓊脂糖凝膠上以低電壓(約5vcm-1)電泳30-60min;
(2)用解剖刀或剃刀片切割含有dna片段的凝膠,盡可能的靠近dna片段切割,以減小凝膠的含量,將膠片放在事先稱重的1.5ml離心管并稱重。記錄膠片的重量。注意避免長時間暴露在紫外燈下,損害dna而影響后續實驗;
(3)加1∶1量的bindingbuffer到膠片中(量以重量計,如每100毫克瓊脂糖凝膠加100微升的bindingbuffer);
(4)在50-60℃的條件下溫育凝膠混合物10min,期間顛倒混勻2-3次,促進膠融化,保證膠全部溶解,在上柱前將凝膠混合物快速渦旋混勻一次;
(5)轉移最多800μl凝膠溶解液到基因回收純化柱,13000g離心1min,棄去流出液,然后將柱放回相同的收集管;
(6)加入700μlwashbuffer(已用乙醇稀釋)到genejet純化柱。13000g離心1min,棄去流出液,然后將柱放回相同的收集管;
(7)離心空genejet純化柱13000g離心1min,徹底去除殘留的washbuffer;
(8)將genejet純化柱轉移到一個干凈的1.5ml離心管,加30-50μlddh2o(可60℃預熱)于純化柱膜,13000g離心1min;
(9)丟掉genejet純化柱并儲存純化的dna在-20℃。
經純化后的pcr產物用限制性內切酶進行雙酶切,采用neb公司t4dna連接酶定向連入經相同雙酶切的表達載體pmal-c2x中(方法如下),連接產物經轉化、藍白菌落篩選及陽性克隆的初步篩選,并送樣測序鑒定,得到經測序核苷酸序列無突變重組質粒pmaltps,并將經的重組質粒pmaltps轉化至大腸桿菌e.colitransb(de3)表達感受態細胞。與此同時,將擬南芥中功能明確的atcps(催化ggpp形成ent-cpp)、atks(催化ent-cpp形成對映貝殼杉烯)及丹參中功能明確的smcps1(催化ggpp形成nor-cpp)、smksl1(催化nor-cpp形成次丹參酮二烯)構建到pmal-c2x原核表達載體上(smcps1、smksl1利用實驗室保存的重組質粒pet32smcps1及pet32smksl1),將雷公藤twcps3、twks分別與其他植物來源的cps、ks進行對此。
采用takaraquickcut酶進行雙酶切反應,反應體系如下(50μl):
內切酶1酶切完成后,加入1μl內切酶2繼續酶切。注意:最適酶切溫度較低的內切酶應先進行酶切,若最適酶切溫度相同,可同時加入同時酶切。
采用nebt4dna快速連接試劑盒進行dna片段連接反應,連接反應體系如下:
連接反應(20μl體系)
*dna與載體摩爾此約為3:1-10:1
25℃連接5min(可適當延長連接時間)。
表1構建原核表達載體引物序列
實施例二、重組蛋白誘導表達
誘導表達
100mmol·l-1iptg:稱取238.3mg的iptg用10ml的ddh2o溶解,過濾分裝-20℃保存;lb培養基:trytone1.0%,yeastextract0.5%,nacl1.0%,agar1.5%,ph7.0。
操作步驟:
(1)將酶切鑒定正確且經測序驗證的含目的基因的重組質粒,取1μl轉化至50μltransb(de3)感受態細胞中,涂板lb+amp(氨芐青霉素鈉)固體平板,37℃倒置培養12-16h;
(2)挑取單克隆菌落經酶切鑒定后轉到含100μg·ml-1amp的2mllb液體培養基中,37℃振蕩培養至od600到0.6-1.0;
(3)取1ml菌液5000g4℃離心5min收集菌體,用新鮮lb+amp液體培養基混懸菌體,轉接到100mllb+amp液體培養基中;
(4)37℃培養至宿主菌密度(od600)達到0.6-1.0時,加入適量iptg誘導劑(終濃度約0.4mm),在低溫下(16℃)誘導培養8h;
(5)4℃3000g離心20min收獲菌體,預冷的5mlhepes緩沖液(50mmhepes,100mmkcl,7.5mmmgcl2,5mmdtt,1mmpmsf,5%甘油,ph7.2)重懸;
(6)置冰浴中超聲破菌(30%功率,超聲5s,間隔5s,持續3min);大腸桿菌破碎液在4℃,15000g離心30min,取上清液;
(7)上清液經蛋白超濾管濃縮(4℃5000g40min)至1.5ml。
取上清進行十二烷基硫酸鈉-聚丙烯酰胺(sds-page)凝膠電泳檢測。
以空質粒pmal-c2x轉化transb(de3)表達菌進行表達為對照。
實施例三、十二烷基硫酸鈉-聚丙烯酰胺凝膠電泳(sds-page)(圖2)
試劑配制
30%丙烯酰胺貯存液(神經毒性,操作時配戴口罩及手套):在通風櫥中,稱取丙烯酰胺29.2g,甲叉雙丙烯酰胺0.8g,加ddh2o溶解后,定容到100ml。針頭濾器過濾后置棕色瓶中,4℃保存;
ph8.8tris-hcl分離膠緩沖液:配制1.5mtris-hcl,并調節ph至8.8,4℃保存;
ph6.8tris-hcl濃縮膠緩沖液:配制1mtris-hcl,并調節ph至6.8,4℃保存;
10%sds:稱取sds1.0g,蒸餾水10ml溶解,4℃保存;
10%過硫酸銨(aps):取aps1.0g,蒸餾水10ml溶解,4℃保存;
temed(四乙基乙二胺)原液;
5×樣品緩沖液(10ml):0.6ml1mol·l-1的tris-hcl(ph6.8),5ml50%甘油,2ml10%的sds,0.5ml巰基乙醇,1ml1%溴酚藍,0.9ml蒸餾水。可在4℃保存數周,或在-20℃保存數月;
10×電泳緩沖液:稱取tris30.38g,甘氨酸144g,sds10.8g,加蒸餾水約900ml,調ph8.3后,用蒸餾水定容至1000ml,置4℃保存,臨用前稀釋10倍。
實施例四、樣品制備
將蛋白質樣品與5×樣品緩沖液在一個eppendorf管中混合,放入100℃加熱5-10min,常溫12000g離心3min,取上清點樣。
實施例五、電泳
操作步驟
(1)將玻璃板、樣品梳用洗滌劑洗凈,用ddh2o沖洗數次,晾干;
(2)安裝玻璃板;
(3)按如下體積配制10%分離膠15ml(配制兩塊膠),混勻;
(4)向玻璃板間灌制分離膠,立即加入1mlddh2o壓平膠面,大約20min后膠即可聚合;
(5)按如下體積配制5%濃縮膠5ml(配制兩塊膠),混勻;
(6)將上層ddh2o傾去,濾紙吸干,灌制濃縮膠,插入樣品梳;
(7)裝好電泳系統,加入電泳緩沖液,上樣5-10μl;
(8)恒流40ma,溴酚藍跑出分離膠30min后,停止電泳;
(9)卸下膠板,剝離膠,染色,照膠。
實施例六、考馬斯亮藍染色
試劑配制
考馬斯亮藍r250染色液:0.25%考馬斯亮藍r250(w/v),45%甲醇(v/v),10%冰乙酸;
脫色液(1000ml):100ml冰乙酸,250ml乙醇,蒸餾水補足至1000ml。
染色步驟
室溫染色45min-60min(或微波爐快染,高火20s,兩次);用蒸餾水清洗3-5遍;加入脫色液,置于100rpm搖床上脫色,每20min更換一次脫色液至膠透明,脫色完成后,用umaxpowerlook2100xl掃描儀進行照膠。
實施例七、酶促反應
以ggpp為底物的酶促反應
取實施例二、步驟(7)獲得的濃縮重組cps蛋白上清進行酶促反應。并以pmal-c2x空載體、pmal-c2x-atcps、pet32a(+)-smcps1轉化大腸桿菌transb(de3)表達感受態細胞,所破碎濃縮得到的重組蛋白上清進行酶促反應作為對照。
酶促反應體系如下:
濃縮重組蛋白上清182μl
底物ggpp(200μm)18μl
反應產物充分混合(槍頭吹打),在室溫(25℃)、黑暗的環境下反應2h;反應結束后用正己烷抽提3次,每次加入0.5ml,棄有機相,留水相(留一層正己烷覆蓋水相,防止產物氧化);然后用n2將水相中的正己烷徹底吹干,以免影響下一步的脫磷反應。
脫磷反應體系如下:
反應產物充分混合(槍頭吹打),在37℃下反應4h;脫磷后的產物再用正己烷抽提3次,每次加入0.5ml,將抽提所得有機相匯合在一起;用n2將提取液吹干,并加入60μl正己烷溶解,用于gc-ms分析。
以cpp為底物的酶促反應
對twks進行功能分析的酶促反應是以經cps酶催化ggpp所產生的產物(cpp)作為反應底物,分析twks酶催化所得產物的化學結構,從而鑒定twks功能。具體實驗過程如下:
(1)配制如下酶促反應體系,在室溫(25℃)黑暗條件下反應2小時,使ggpp充分轉化成cpp;
濃縮重組ks蛋白上清182μl
底物ggpp(200μm)18μl
(2)向上述反應混合液中加入等體積的twks酶,并補充mgcl2至10mm,在室溫(25℃)黑暗條件下,反應過夜(12-16h);
(3)反應結束后,體系用正己烷抽提3次(每次0.5ml),所得正己烷相用n2吹干,然后加入60μl正己烷溶解進行gc-ms分析。
實施例八、反應產物gc-ms檢測
gc-ms分析條件為:取1μl的進樣,無分流的模式下,50℃保持2min,20℃·min-1升至300℃,保持20min;進樣口溫度250℃,離子源溫度250℃,電子能量70ev,對樣品進行20-650m/z范圍掃描。gc-ms儀器為thermoscientific公司thermotrace1310/tsq8000gaschromatograph,色譜柱為db-5ms(30m×0.25mm)。(圖5、6)
實施例九、定點突變
采用全式金試劑盒fastmutagenesissystem對twcps2、twks進行定點突變操作。
定點突變引物設計
除突變位點外,兩條引物長度大約25-30bp,5’端重疊區包含15-20bp,3’端延伸區包含至少10bp;突變位點位于兩條引物上,分別位于正向突變引物重疊區下游、緊鄰重疊區,反向突變引物5’端(見圖3)。
根據以上原則設計定點突變引物,在‘dxdd’功能結構域處將twcps2dtdc(311)突變成dtdd(311),twcps2c311d即,將twksdadt(318)突變成dadd(318),即twkst318d;在twks第608位氨基酸處,將氨基酸a突變成m,即twksa608m,定點突變引物序列見表2。
表2定點突變引物序列
定點突變反應
定點突變反應體系:
(1)pcr反應條件:
(2)取10μlpcr產物,1.0%瓊脂糖凝膠電泳檢測。即使觀察到多條擴增條帶,如果目的條帶大小正確,可繼續用dmt消化及轉化反應;
(3)加1μldmt酶于pcr產物中,混勻,37℃孵育1h;
(4)加入2-5μldmt酶消化產物于50μldmt感受態細胞中,輕輕混勻,冰浴30min;
(5)42℃準確熱激45s,立即置于冰上2min;
(6)加入250μl平衡至室溫的lb培養基,37℃225rpm培養1h;
(7)取200μl菌液涂板,37℃倒置培養過夜。
(8)藍白菌落篩選及陽性克隆的初步篩選,方法同2.2.3.1.4,并送樣測序驗證能突變是否成功。
實施例十、酵母發酵
雷公藤二萜合酶真核表達載體構建
采用雙酶切方法分別將二萜合酶基因twcps1、twges2及組合twcps3+twks、twcps3+twksa608m構建到真核表達載體pesc-trp上,方法同3.2.1。先將twcps1、twges2、twks、twksa608mpcr產物(純化)及載體質粒pesc-trp用限制性內切酶進行雙酶切,采用neb公司t4dna連接酶定向連接,連接產物經轉化、藍白菌落篩選及陽性克隆的初步篩選,并送樣測序鑒定,得到經測序核苷酸序列無突變重組質粒pesctwcps1、pesctwges2、pesctwks及pesctwksa608m,再將twcps3pcr產物(純化)及重組質粒pesctwks、pesctwksa608m用限制性內切酶進行雙酶切,采用neb公司t4dna連接酶定向連接,連接產物經轉化、藍白菌落篩選及陽性克隆的初步篩選,并送樣測序鑒定,得到經測序核苷酸序列無突變重組質粒pesc-(twcps3+twks)、pesc-(twcps3+twksa608m)。見圖4,引物序列見表3。
表3構建真核表達載體引物序列
酵母感受態制備
sd-his固體平板:sd-his+2%葡萄糖+4%agar;不加agar則成為相應液體培養基;sd-his-trp固體平板:sd-his-trp+2%葡萄糖+4%agar;不加agar則成為相應液體培養基。
by-t20酵母菌是在缺陷型酵母菌株by4741(基因型:matahis3δ1leu2δ0met15δ0ura3δ0)基礎上進行改造,致使其高效合成ggpp,并能回補his,故將by-t20酵母菌涂布sd-his(缺his)固體平板,30℃倒置培養72h。
采用zymoresearchfrozen-ezyeasttransformationii試劑盒做酵母感受態細胞:
(1)從sd-his平板上挑取新活化的單菌落,接種于10mlsd-his液體培養基中,30℃下振蕩培養至od600=0.8-1.0左右;
(2)室溫,500g離心4min,去上清;
(3)加入10mlfrozen-ezsolution1懸浮菌體,室溫,500g離心4min,去上清;
(4)加入1mlfrozen-ezsolution2懸浮菌體,分裝至滅菌的1.5mlep管中,每管50μl;
(5)禁止用液氮速凍感受態細胞,應緩慢降溫至-70℃(4℃,1h;-20℃,1h;-40℃,1h;-70℃保存)。
轉化
(1)取0.2-1μg重組質粒(少于5μl)與50μl感受態細胞混合;
(2)加入500μlfrozen-ezsolution3,劇烈混勻;
(3)30℃孵育45min,期間在此混勻2-3次;
(4)取50-150μl孵育的菌液,涂布相應缺陷型sd平板(sd-his-trp),晾干后,置于30℃倒置培養48-96h。
發酵
酵母質粒提取及鑒定
挑取sd-his-trp固體平板上長出來的單菌落,置于10mlsd-his-trp液體培養基中,30℃250rpm48h;提取質粒,用特異性引物pcr驗證重組質粒是否轉入by-t20酵母菌。
酵母質粒提取(天根公司酵母質粒小提試劑盒):
(1)柱平衡:向吸附柱cp2中(吸附柱放入收集管中)加入500μl的平衡液bl,12000rpm離心1min,倒掉收集管中的廢液,將吸附柱重新放回收集管中(當天處理當天使用);
(2)取1-5ml酵母培養物,12000rpm離心1min,盡量吸除上清(菌液較多時可以通過多次離心將菌體收集到一個離心管中);
(3)向菌體中加入250μl溶液yp1(已加入rnasea)重懸沉淀,徹底懸浮菌體,加入直徑為0.45-0.55mm的酸洗玻璃珠,渦旋振蕩10min;
(4)向管中加入250μl溶液yp2,溫和地上下翻轉6-8次,使菌體充分混勻,室溫放置5-10min(避免劇烈震蕩,以免污染基因組dna);
(5)向管中加入350μl溶液yp3,立即溫和地上下翻轉6-8次,充分混勻,此時會出現白色絮狀沉淀,12000rpm離心20min;取上清部分,再次離心12000rpm離心20min,以得到無微小白色沉淀的上清液;
(6)小心將上清液加入吸附柱cp2中(吸附柱放入收集管中),12000rpm離心1min,倒掉廢液,將吸附柱cp2放入收集管中;
(7)向吸附柱cp2中加入500μl緩沖液pd,12000rpm離心1min,倒掉廢液;
(8)向吸附柱cp2中加入600μl漂洗液(已加入無水乙醇),12000rpm離心1min,倒掉廢液,將吸附柱cp2放入收集管中;
(9)重復步驟8;
(10)將吸附柱cp2放入收集管中置于12000rpm離心2min,去除殘余的漂洗液;
(11)將吸附柱cp2置于一個干凈的1.5mlep管中,向吸附膜的中間部位滴加50-100μlddh2o,室溫放置2min,12000rpm離心2min將質粒溶液收集到ep管中,保存-20℃。
所提取得質粒用于pcr鑒定重組質粒是否轉入by-t20酵母菌中。
pcr反應體系:
pcr反應條件:
(備注:*表示pcr產物的片段>3kb,且每加1kb則延伸溫度延長1min)
1.0%瓊脂糖凝膠電泳檢測,若出現目的片段條帶,則可以證明重組質粒成功轉入菌體。
半乳糖誘導發酵
誘導型sd-his-trp液體培養基:sd-his-trp+2%半乳糖(過濾除菌)
(1)將pcr鑒定的陽性菌液(即重組質粒成功轉入的by-t20酵母菌),轉接入50mlsd-his-trp液體培養基中,30℃250rpm培養12-16h;
(2)室溫5000g5min離心收集菌體,轉接到100ml誘導型sd-his-trp液體培養基中,30℃250rpm誘導72h;
發酵產物提取
目標成分為萜類化合物,脂溶性,易溶于正己烷,因此選取正己烷為溶劑萃取目標萜類化合物。萃取步驟如下:
(1)收集發酵完成菌液,加入等體積的正己烷;
(2)超聲破菌1h,期間多次振蕩混搖;
(3)分液漏斗萃取兩次;混合兩次的萃取液,加入適量的無水硫酸鈉(120℃烘干30min),邊加邊搖,出去萃取液的水分;
(4)旋轉蒸發儀上濃縮至近干,
(5)吸取濃縮液,過0.22μmptfe針頭濾器,過濾液儲存于液相小瓶中,封口膜密閉,保存于4℃冰箱。
提取物gc-ms檢測
gc-ms分析條件為:取1μl的進樣,無分流的模式下,50℃保持2min,20℃·min-1升至300℃,保持20min;進樣口溫度250℃,離子源溫度250℃,電子能量70ev,對樣品進行20-650m/z范圍掃描。gc-ms儀器為thermoscientific公司thermotrace1310/tsq8000gaschromatograph,色譜柱為db-5ms(30m×0.25mm)。(圖7、8、9)
上述說明并非對本發明的限制,本發明也并不限于上述舉例。本技術領域的普通技術人員在本發明的實質范圍內,作出的變化、改型、添加或替換,也應屬于本發明的保護范圍,本發明的保護范圍以權利要求書為準。
sequencelisting
<110>首都醫科大學
<120>雷公藤twks與twcps3在制備貝殼杉烷型二萜化合物中的應用
<130>無
<160>28
<170>patentinversion3.5
<210>1
<211>816
<212>prt
<213>雷公藤
<400>1
metvalilemetserserhisglnilepheservalserserserser
151015
tyrleuhisproproleuleupheproglyleusersertyrthrthr
202530
lysasplysargvalcystyrpheaspserthrargleuilecysarg
354045
alaileserlysproalaargthrthrproglutyrserglyvalleu
505560
glnasnglyleuproleuilelystrparggluilevalgluaspasp
65707580
ileglngluglnglugluproleulysvalserleugluasnglnile
859095
argglnglyvalaspilevallyssermetleuglysermetgluasp
100105110
glygluileserileseralatyraspthralatrpvalalaleuval
115120125
gluasnilehishisprothrglyserproglnpheproserthrleu
130135140
glntrpilealaasnasnglnleuproaspglysertrpglyasppro
145150155160
tyrvalpheleualahisaspargleuileasnthrleualacysval
165170175
ilealaleulyslystrpasnilehisproasnlystyrlyslysgly
180185190
leuserphevallysgluasnileserlysleuglulysgluasnglu
195200205
gluhismetproileglyphegluilealapheproserleuleuglu
210215220
metalalyslysleuglyilegluileproaspaspcysproalaleu
225230235240
glnaspiletyralalysargaspleulysleuthrargileprolys
245250255
aspilemethisasnvalprothrthrleuleuhisserleuglugly
260265270
leuproglyleuasptrpglulysleuvalargleuglncyslysasp
275280285
glyserpheleupheserproserserthralacysalaleumethis
290295300
thrlysaspglyasncysphesertyrleuasnasnmetvalglnlys
305310315320
pheasnglyglyvalproasnvaltyrprovalaspleuphegluhis
325330335
iletrpservalaspargleuleuargleuglyileserargphephe
340345350
glnprogluilelysglncysleuasphisvalhisargtyrtrpthr
355360365
lysaspglyilecystrpalaargasnserhisvalglnaspileasp
370375380
aspthrsermetglypheargleuleuargleuhisglytyrgluval
385390395400
serproaspvalphelysglnphearglysglyaspgluphevalcys
405410415
phealaglyglnserasnglnalailethrglyiletyrasnleutyr
420425430
argalaserglnleumetpheproglugluproileleuvalgluala
435440445
lyslysphealaargasnpheleuargglulysargalavalasnglu
450455460
leuleuasplystrpileilethrlysaspleuproglygluvalgly
465470475480
phealaleuaspvalprotrptyralacysleuproargvalgluthr
485490495
argleutyrilegluglntyrglyglyglnaspaspvaltrpilegly
500505510
lysserleutyrargmetprotyrvalasnasnasniletyrleuglu
515520525
leualalysleuasptyrasnasncysglnserleuhisargthrglu
530535540
trpaspasnileglnglutrptyrgluglutyrasnvalglyglyphe
545550555560
glyvalserlysargserleuleulysthrtyrphevalalathrala
565570575
serilephegluprogluargservalgluargleuvaltrpthrlys
580585590
thralaileleuleuglualailealasertyrphelysasnserarg
595600605
glugluargilegluphevalasnglupheglnlyspheprolysthr
610615620
argglytyrilelysglyargargleuaspglylysmetalathrlys
625630635640
gluvalileglumetvalphealaalaleuasnhispheserleuasp
645650655
serleuvalvalhisglyglnaspilethrhishisleutyrglnser
660665670
trpvallystrpvalleuthrtrpglnglugluglyaspargargglu
675680685
glyglualagluleuleuvalglnthrileasnleumetalaglyhis
690695700
thrproserglngluleuleutyrgluargleuphelysleuthrasn
705710715720
lysvalcyshisglnleuglyhistyrhishisleuasnlysasplys
725730735
glnproglnglnvalgluaspasnglyglytyrasnasnserasnpro
740745750
gluserileserlysleuglnilegluseraspmetarggluleuval
755760765
glnleuvalleuasnserseraspglymetaspserasnilelysgln
770775780
thrpheleuthrvalthrlysserphetyrtyrasnalaphethrhis
785790795800
proglythrvalasntyrhisilealalysvalleuphegluargval
805810815
<210>2
<211>2748
<212>dna
<213>雷公藤
<400>2
acacagagttagacaaagtgaatagttttggtttaggatgatcccatgcacctattagcg60
ggaatctcctgggaagtcgtttctgtcttgttgccctggcccgccacctcctccctctta120
ttacttttgaatccttttaagccccctttttgctctgtctctctctctctctctgtgtct180
ctgcaaataaacattaacacaaatccaatcaatcatggtgatcatgtcctctcatcaaat240
cttctctgtttcttcctcctcctaccttcaccctcctctgcttttcccaggattaagttc300
ctacactactaaagacaaacgagtctgctactttgatagcactcgcttaatctgccgtgc360
aatatccaagcctgctcgcacaactccagaatactcgggcgtgcttcaaaacggtctgcc420
attgataaagtggcgtgagattgtggaggatgacatacaagaacaagaagaacctcttaa480
ggtttccttggaaaaccagattagacaaggtgtcgacattgttaagtcgatgttgggatc540
aatggaggatggcgagattagcatttcagcttatgacactgcatgggttgcacttgttga600
aaacattcaccacccaactggttctcctcaattcccatccactcttcaatggattgccaa660
caatcagctccccgatggctcctggggtgatccctacgtgttcttggcacacgatcgatt720
aatcaatacgttagcttgtgttatcgctctcaagaaatggaatattcaccccaacaaata780
caaaaaaggattgtcgtttgtgaaagagaacataagcaagctagaaaaggagaatgagga840
gcacatgccaataggatttgaaatcgctttcccatcacttcttgagatggctaaaaaact900
agggattgaaatacctgatgattgtccagccttgcaagacatctatgccaagagagatct960
caaactcacaaggataccaaaggacataatgcataatgtgcccacaacactactccatag1020
cttggaaggattgccaggcctggattgggaaaagcttgtaagattgcaatgcaaagatgg1080
gtcattcttgttctctccatcttccactgcctgtgcactcatgcacactaaagatggcaa1140
ttgcttctcctacttgaacaacatggtacaaaagttcaatggaggagtaccgaatgtcta1200
cccggttgacctgttcgaacatatttggtctgttgatcgcttgctacgcctaggaatctc1260
cagattttttcagccagaaatcaaacaatgtcttgatcatgttcacagatattggaccaa1320
agacggaatttgttgggcaagaaattcgcatgttcaagatattgatgacacatcaatggg1380
attcaggttgcttagattgcatggatacgaggtttctcctgacgtgtttaagcaatttag1440
aaaaggcgacgagttcgtttgtttcgcgggacaatcaaatcaggcgattactggaatcta1500
taacctgtatagggcttctcaattgatgttccctgaagagcctattcttgtggaagccaa1560
aaaatttgcaagaaatttcttgagggaaaaaagagctgttaatgaactactagataaatg1620
gatcataaccaaagatttaccaggcgaggtggggtttgccttggatgttccatggtacgc1680
atgcttgcctagagttgagacgaggctttacattgaacaatacggtggccaagatgacgt1740
atggattggcaagtctctttacaggatgccctatgtcaacaacaatatatatctggagct1800
agcaaagctagactacaacaattgccaatccttgcatcgcactgaatgggacaatatcca1860
agagtggtacgaagaatacaatgttggaggatttggggtcagcaaaaggagccttcttaa1920
gacatattttgtagctacagccagtatttttgaacccgaaagatcggtagagcgactcgt1980
ttggacaaagactgcaatcttgttagaagctattgcatcttattttaaaaattctagaga2040
agaaaggattgaatttgtaaatgaattccaaaaatttcccaagacaagaggttacattaa2100
aggaaggagattagatggaaagatggcaacaaaagaagttattgagatggtgtttgcagc2160
actaaatcatttttcattggattcattggtggtgcatggtcaagacattactcaccattt2220
atatcaatcctgggtaaagtgggtgctgacgtggcaagaggaaggagacagaagagaagg2280
agaagcagagctattggtgcagacaataaacttgatggctggccacacaccctctcagga2340
gttgttgtatgagcgactcttcaaactcacaaacaaggtttgccatcagcttggtcacta2400
tcaccacctcaacaaagacaaacaacctcaacaggtagaagacaatggaggatacaataa2460
ttcaaaccctgaaagcatttcaaagctccaaatagagagtgacatgagagagctagtgca2520
gctggtactaaattcttcagatggcatggactctaatatcaagcaaacattcttaacagt2580
cacaaagagcttttactacaatgctttcactcatcctgggactgtcaactaccatattgc2640
caaagtactctttgaaagagtgtaagattagttgacatttgaatattgaaaataatgttg2700
aggtcattgtagataaccaaaaaaaaaaaaaaaaaaaaaaaaaaaaaa2748
<210>3
<211>781
<212>prt
<213>雷公藤
<400>3
metmetserleuserhisproasncysilearghisserserleupro
151015
ileseralathrleuprolysserlyssergluvalleuthrgluthr
202530
aspalathrileleutyrpheglngluthrlysgluargilelyslys
354045
metpheasplysalagluleuservalseralatyraspthralatrp
505560
valalametvalserserproasnserhisglnalaprocysphepro
65707580
glucysvalasntrpleuleuaspasnglnleuseraspglysertrp
859095
glyleuproprohishisproserleuvallysaspalaleuserser
100105110
thrleualacysleuleualaleulysargtrpglyleuglyglugln
115120125
glnmetthrlysglyleuglnpheilegluserasnphethrserile
130135140
asnaspglugluglnhisthrproileglypheasnileilephepro
145150155160
glymetilegluthralaileaspmetasnleuasnleuproleuarg
165170175
sergluaspileasnvalmetleuhisasnargaspleugluleuarg
180185190
argasnlysphegluglyargglualatyrleualatyrvalserglu
195200205
glymetglylysleuglnasptrpglumetvalmetlystyrglnarg
210215220
lysasnglyserleupheasnserproserthrthralaalaalaleu
225230235240
serglnleuglyasnalaglycysphehistyrileasnserleuile
245250255
alalyspheglyasnalavalprothrvaltyrproserasplystyr
260265270
alaleuleucysmetilegluserleugluargleuglyileaspser
275280285
hispheserglngluileargaspvalleuglugluthrtyrargcys
290295300
trpleuglnglyaspglugluilepheseraspalaaspthrcysala
305310315320
metalapheargileleuargvalhisglytyrgluvalserserasp
325330335
proleuthrglntyralagluhishispheserargserpheglygly
340345350
hisleulysasppheserthralaleugluleuphelysalasergln
355360365
phevalilepheproglugluserglyleuglulysglnmetsertrp
370375380
thrasnglnpheleulysglnglupheserasnglythrthrargala
385390395400
aspargphearglystyrpheserilegluvalhisaspthrleulys
405410415
pheprophehisalaasnvalgluargleualahisargargasnile
420425430
gluhishishisvalaspasnthrargileleulysthrsertyrcys
435440445
pheserasnileserasnalaasppheleuglnleualavalgluasp
450455460
pheasnargcysglnserilehisargglugluleulyshisleuglu
465470475480
argtrpvalvalgluthrlysleuaspargleulysphealaarggln
485490495
lysmetalatyrcystyrpheseralaalaglythrcyspheserpro
500505510
gluleuseraspalaargilesertrpalalysasnservalleuthr
515520525
thrvalalaaspaspphepheaspilevalglysergluglugluleu
530535540
alaasnleuvalhisleuleugluasntrpaspalaasnglyserpro
545550555560
histyrcyssergluprovalgluileilepheseralaleuargser
565570575
thrilecysgluileglyasplysalaleualatrpglnglyargser
580585590
valthrhishisvalileglumettrpleuaspleuleulysserala
595600605
leuargglualaglutrpalaargasnlysvalvalprothrpheasp
610615620
glutyrvalgluasnglytyrvalsermetalaleuglyproileval
625630635640
leuproalavaltyrleuileglyprolysvalserglugluvalval
645650655
argserprogluphehisasnleuphelysleumetserilecysgly
660665670
argleuileasnaspthrargthrphelysarggluserglualagly
675680685
lysleuasnservalleuleuhismetilehisserglyserglythr
690695700
thrglugluglualavalglulysileargglyleuilealaaspgly
705710715720
glnarggluleuleuargleuvalleuglnglulysaspserileval
725730735
proargalacyslysaspleuphetrplysmetvalglnvalleuhis
740745750
leuphetyrmetaspglyaspglypheserserproaspmetmetleu
755760765
asnalavalasnalaleuilearggluproileserleu
770775780
<210>4
<211>2548
<212>dna
<213>雷公藤
<400>4
aagtctggtgcatgatttgaagaaaattattatttgggcagagaggggactctactgacc60
actgagtaatattcttcatatatagcttgtttgggattctggtgccatgatgtctctatc120
gcatcccaattgtattaggcactcctctttaccaatatcagcaaccttaccgaagagtaa180
atcagaagtactgaccgaaaccgatgccaccattctgtattttcaagagactaaagaaag240
aattaagaagatgtttgataaggccgaactctctgtttcggcatatgacaccgcttgggt300
agcaatggtatcatctcctaattcccatcaggccccttgcttccctgagtgcgtaaattg360
gttgttggataatcaactctctgatggttcttggggtcttcctcctcatcatccctcgtt420
agttaaagatgctttgtcatccactttagcatgtctccttgctttgaagcgatggggtct480
tggtgaacaacaaatgacaaaaggactccaatttattgaatcaaatttcacttcaattaa540
tgatgaggagcaacacaccccaattggattcaatataatttttcctgggatgattgagac600
tgctatagatatgaatttgaatcttcctctaagatcggaagacataaatgtgatgcttca660
caatagagacttggagcttaggagaaataaattcgagggaagggaagcgtacttagccta720
tgtttccgaaggaatgggaaaattacaggattgggaaatggttatgaaatatcagagaaa780
gaatgggtcactgttcaattcaccgtctactactgcagctgctcttagccaacttggaaa840
tgctggttgttttcattacatcaactcacttatagcaaagtttgggaatgcagttccgac900
agtttatccttctgataaatatgctcttctttgtatgattgagagtcttgagaggctggg960
aattgatagtcatttcagtcaggaaattagagatgtactggaggaaacctacagatgctg1020
gttgcagggagatgaagagatattttccgatgctgacacatgtgccatggcatttcgaat1080
attacgtgtacatggatatgaagtctcttcagatccattaactcaatatgctgaacatca1140
tttctcccgttcttttggaggccatttgaaggattttagtactgccttggagttattcaa1200
agcttcacaatttgtaatattcccagaggaatcaggtctcgaaaaacagatgtcgtggac1260
aaatcaatttctaaagcaggaattctctaatgggacaactcgtgcagatagattccgcaa1320
atattttagcatagaagtacatgatactcttaagtttcccttccatgcaaatgtggaacg1380
cttagcacacaggagaaatatagagcatcaccatgtagataacacaagaatattaaagac1440
ttcatattgtttttcaaatatcagcaacgctgattttctgcaactggcggtggaagactt1500
caataggtgccaatcaatacaccgcgaagaactcaaacatcttgagaggtgggttgtgga1560
gaccaaattagacaggctaaagtttgctagacagaaaatggcatactgttacttttctgc1620
tgctggaacatgcttctctcctgaactatcagatgctcgcatctcatgggccaaaaacag1680
tgtgcttacaactgttgccgacgacttctttgacattgtggggtcggaagaggaactggc1740
aaaccttgtacacttgcttgagaattgggatgcaaatggtagccctcattattgttctga1800
gccagttgaaatcatattttcagcacttcgtagcacaatttgtgagatcggagacaaagc1860
attagcatggcaagggagaagtgtgacacaccatgttattgagatgtggttggatttgct1920
aaagtctgcgttaagggaagccgagtgggcgagaaacaaagtagtgccaacattcgatga1980
atacgtggaaaatggatatgtgtctatggccttgggaccaattgttcttccagcagtcta2040
tctcatcgggcctaaagtttcagaggaggttgtccgaagccctgaattccataacctctt2100
caaacttatgagcatttgtggacgtctaatcaatgacacccgcacatttaagagggaatc2160
cgaggctgggaaattgaattctgttttgttgcacatgatccacagtggtagtggaacaac2220
ggaggaagaagctgttgaaaagattagaggtttgattgcggatggacaaagagaactgct2280
aaggctagtgttgcaggagaaagacagcatagttcctagagcttgcaaggacttgttctg2340
gaaaatggtccaagtgttgcacctattttacatggatggagatggattctcttcacctga2400
catgatgctcaatgcagtaaacgctttgatacgtgaacccatctctctataattgcaggc2460
tctaccctttacccattgaaaggattttacaataaattgggatggattgtgtgttttcag2520
ccaaaaaaaaaaaaaaaaaaaaaaaaaa2548
<210>5
<211>33
<212>dna
<213>人工序列
<400>5
cgcggatccatggtgatcatgtcctctcatcaa33
<210>6
<211>36
<212>dna
<213>人工序列
<400>6
gcactgcagttacactctttcaaagagtactttggc36
<210>7
<211>35
<212>dna
<213>人工序列
<400>7
cgcggatccatgatgtctctatcgcatcccaattg35
<210>8
<211>39
<212>dna
<213>人工序列
<400>8
cgcgtcgacttatagagagatgggttcacgtatcaaagc39
<210>9
<211>39
<212>dna
<213>人工序列
<400>9
cgcggatccatgtctcttcagtatcatgttctaaactcc39
<210>10
<211>37
<212>dna
<213>人工序列
<400>10
gcactgcagctagactttttgaaacaagactttggag37
<210>11
<211>32
<212>dna
<213>人工序列
<400>11
cgcggatccatgtctatcaaccttcgctcctc32
<210>12
<211>35
<212>dna
<213>人工序列
<400>12
cgcgtcgactcaagttaaagattcttcctgtaagc35
<210>13
<211>32
<212>dna
<213>人工序列
<400>13
cgcagcttcgcgataccgatgataccgccatg32
<210>14
<211>36
<212>dna
<213>人工序列
<400>14
tcatcggtatcgcgaagctgcgaatttcttgtccag36
<210>15
<211>35
<212>dna
<213>人工序列
<400>15
gagatattttccgatgctgacgattgtgccatggc35
<210>16
<211>37
<212>dna
<213>人工序列
<400>16
atcgtcagcatcggaaaatatctcttcatctccctgc37
<210>17
<211>32
<212>dna
<213>人工序列
<400>17
gttggatttgctaaagtctatgttaagggaag32
<210>18
<211>32
<212>dna
<213>人工序列
<400>18
atagactttagcaaatccaaccacatctcaat32
<210>19
<211>35
<212>dna
<213>人工序列
<400>19
cgcggatccgatgcatagtttgttaatgaaaaagg35
<210>20
<211>36
<212>dna
<213>人工序列
<400>20
tcggggcccaaatctactctctcaaagagtactttg36
<210>21
<211>37
<212>dna
<213>人工序列
<400>21
cgcggatccgatggtgatcatgtcctctcatcaaatc37
<210>22
<211>35
<212>dna
<213>人工序列
<400>22
tcggggcccaacactctttcaaagagtactttggc35
<210>23
<211>37
<212>dna
<213>人工序列
<400>23
atttgcggccgcaatgatgtctctatcgcatcccaat37
<210>24
<211>37
<212>dna
<213>人工序列
<400>24
cccatcgatactagagagatgggttcacgtatcaaag37
<210>25
<211>37
<212>dna
<213>人工序列
<400>25
atttgcggccgcaatgatgtctctatcgcatcccaat37
<210>26
<211>37
<212>dna
<213>人工序列
<400>26
cccatcgatactagagagatgggttcacgtatcaaag37
<210>27
<211>34
<212>dna
<213>人工序列
<400>27
cgcggatccgatggatttttcagattcctcaatt34
<210>28
<211>35
<212>dna
<213>人工序列
<400>28
tcggggcccaaaatgaaacatggtgagaattttgg35
- 還沒有人留言評論。精彩留言會獲得點贊!