快速鑒定光皮梾木種群的分子檢測方法與流程
本發明屬于分子生物學技術領域,更具體地,本發明涉及一種快速鑒定光皮梾木種群的分子檢測方法。
背景技術:
光皮梾木(cornuswilsonianawanger)又名光皮樹、油樹、狗骨木、斑皮抽水樹等,屬山茱萸科梾木屬,是我國特有的多用途鄉土木本油料樹種。光皮梾木果油作為食用油已有100年歷史,1981年經國家糧食部鑒定,光皮梾木油為一級食用油;2013年12月,國家衛生和計劃生育委員會批準光皮梾木果實油為我國的新食品原料。動物和臨床試驗結果表明光皮梾木油可顯著降低體內膽固醇,防治高血脂癥,尤其是老年高血脂癥,同時具有減肥功效。光皮梾木是一種兼有經濟、生態和社會效益的重要油料樹種。
但是,一般從表型特征很難區分光皮梾木的種群,尤其是在上個世紀70-80年代,曾經在一定的區域內進行過人為的廣泛傳播,對于收集、選擇和建立基因庫帶來一定的難度。因此,急需解決判定種群起源的準確方法,為開展光皮梾木的育種提供必要的技術支持。
植物的基因組具有很好的穩定性,基本上不受環境影響,且取材方便。因此,通過對植株在基因組差異性的檢測來追蹤其起源是完全可行的。
dna分子標記是快速鑒定生物基因組差異的重要方法之一,其中微衛星標記具有多態性高、穩定性好、操作便捷等諸多優點,通過對目標植物種群進行微衛星標記的檢測,不僅可以區分在性狀上存在明顯差異的個體,而且能夠有效判別種群水平上的相似性。
目前國內外尚無對光皮梾木種群分子快速鑒定的相關報道。
技術實現要素:
為了克服上述現有技術的缺陷,本發明提供了一種非破壞性取樣下快速鑒定光皮梾木種群的分子檢測方法及其引物對,該鑒定方法準確、省時、省力。
為了實現上述發明目的,本發明采取了以下技術方案:
一種快速鑒定光皮梾木種群的分子檢測方法,包括以下步驟:
(1)、以光皮梾木的dna為模板,分別以seqidno:1~2、seqidno:3~4、seqidno:5~6、seqidno:7~8、seqidno:9~10、seqidno:11~12、seqidno:13~14、seqidno:15~16、seqidno:17~18、seqidno:19~20、seqidno:21~22、seqidno:23~24、seqidno:25~26、seqidno:27~28、以及seqidno:29~30作為引物對,進行pcr擴增,獲得微衛星分子標記數據;
(2)、對上述微衛星分子標記數據進行統計分析,鑒定歸類光皮梾木種群。
在其中一些實施例中,步驟(1)中所述pcr擴增的反應程序為:
步驟s1,94℃下預變性5min;
步驟s2,94℃下變性30s;
步驟s3,57℃下退火30s;
步驟s4,72℃下延伸20s;
步驟s5,將所述步驟s2至所述步驟s4重復35次,且最后一次的所述步驟s4持續10min。
在其中一些實施例中,步驟(1)中所述pcr擴增的反應體系為:dna模板1.0μl、引物對0.2μl、mix10μl,加ddh2o至20μl。
本發明還提供了快速鑒定光皮梾木種群的引物對,采取了以下技術方案:
一種快速鑒定光皮梾木種群的引物對,所述引物對包括seqidno:1~2、seqidno:3~4、seqidno:5~6、seqidno:7~8、seqidno:9~10、seqidno:11~12、seqidno:13~14、seqidno:15~16、seqidno:17~18、seqidno:19~20、seqidno:21~22、seqidno:23~24、seqidno:25~26、seqidno:27~28、以及seqidno:29~30。
本發明通過對目標參試植株進行dna提取,根據光皮梾木15個穩定性好、多態性強的微衛星標記引物對進行dna擴增,獲得分子標記數據后,采用統計分析軟件對分子數據進行分析,從而對光皮梾木種群進行歸類。與現有技術相比,本發明具有以下有益效果:
1、本發明的快速鑒定光皮梾木種群的分子檢測方法在非破壞性取樣的前提下快速獲得基因組信息,工作量少且效率高,同時判別準確率高達95%,具有準確、省時和省力的優勢;
2、本發明的快速鑒定光皮梾木種群的分子檢測方法對于物種種群的快速分子檢測具有重要價值和應用前景。
附圖說明
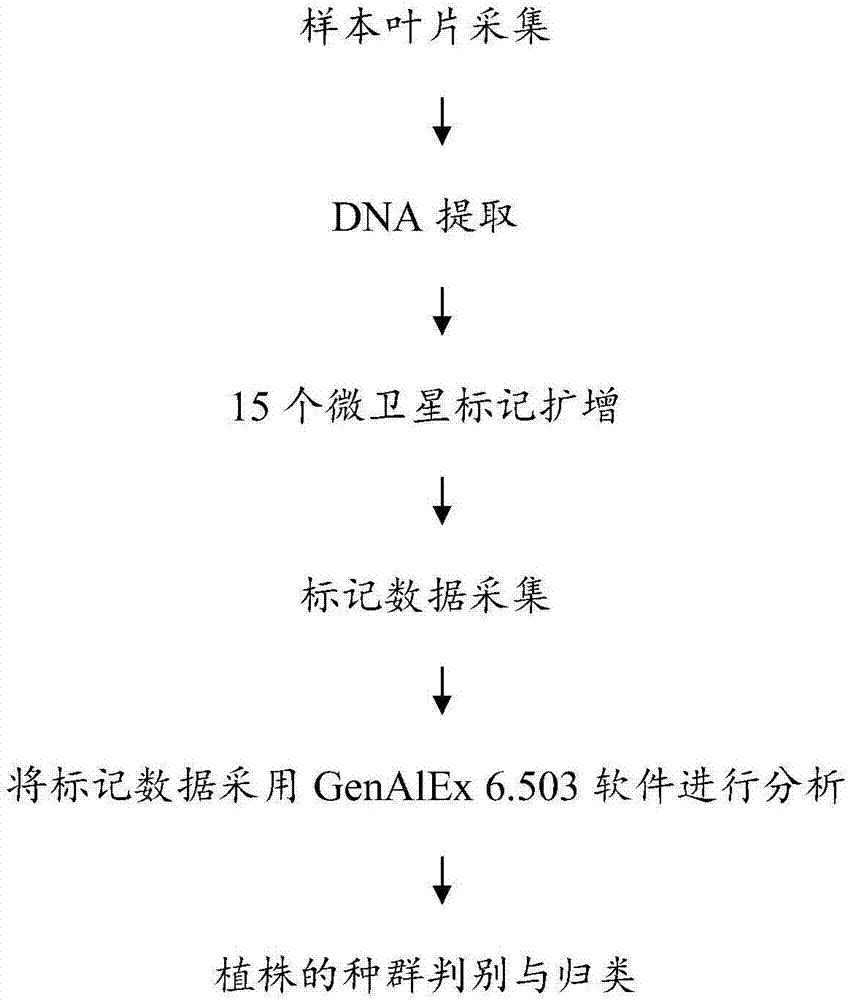
圖1為本發明快速鑒定光皮梾木種群的分子檢測方法的流程圖;
圖2為本發明實施例1中20個光皮梾木植株種群判別二維圖,其中lc:廣東樂昌種群;jx:江西贛州種群。
具體實施方式
下面結合附圖和具體實施例進一步敘述本發明,本發明未述及之處適用于現有技術。下面給出本發明的具體實施例,但實施例僅是為了進一步詳細敘述本發明,并不限制本發明的權利要求。
實施例1一種快速鑒定光皮梾木種群的分子檢測方法
請參閱圖1,為一種快速鑒定光皮梾木種群的分子檢測方法的流程圖,檢測方法包括以下步驟:
(1)、按本領域常規方法對來自江西和廣東2個種群共20個光皮梾木植株的葉片樣品進行dna提取;
(2)、分別以步驟(1)提取的dna為模板,分別以seqidno:1~2、seqidno:3~4、seqidno:5~6、seqidno:7~8、seqidno:9~10、seqidno:11~12、seqidno:13~14、seqidno:15~16、seqidno:17~18、seqidno:19~20、seqidno:21~22、seqidno:23~24、seqidno:25~26、seqidno:27~28、以及seqidno:29~30作為引物對,分別進行pcr擴增,獲得微衛星分子標記數據;
其中,引物對的序列如下:
每個pcr擴增的反應體系(20μl)為:
pcr擴增的反應程序為:
步驟s1,將所述全基因組dna依次在94℃下預變性5min;
步驟s2,將預變性產物在94℃下變性30s,得到變性產物;
步驟s3,將所述變性產物在57℃下退火30s,得到退火產物;
步驟s4,將所述退火產物在72℃下延伸20s;
步驟s5,將所述步驟s2至所述步驟s4重復35次,且最后一次的所述步驟s4持續10min。
(3)、統計分析鑒定歸類光皮梾木種群。
采用非商業化genalex6.503軟件對上述微衛星分子標記數據進行分析,統計分析的基本運算原理:根據同類性質的基因組信息(本實施例中為微衛星標記的dna片段大小)具有相似性的原理,也就是植株的相似度越高,標記的相似性就大,軟件根據每個植株的微衛星標記的特征,計算出植株與植株之間在標記信息上的相似度,根據相似度的大小進行歸類;得到植株的種群判別與歸類。
江西和廣東2個種群共20個植株葉片樣品的統計分析結果如圖2所示,結果表明,20個植株中7株來自廣東種群,13株來自江西種群。
以上所述實施例的各技術特征可以進行任意的組合,為使描述簡潔,未對上述實施例中的各個技術特征所有可能的組合都進行描述,然而,只要這些技術特征的組合不存在矛盾,都應當認為是本說明書記載的范圍。
以上所述實施例僅表達了本發明的幾種實施方式,其描述較為具體和詳細,但并不能因此而理解為對發明專利范圍的限制。應當指出的是,對于本領域的普通技術人員來說,在不脫離本發明構思的前提下,還可以做出若干變形和改進,這些都屬于本發明的保護范圍。因此,本發明專利的保護范圍應以所附權利要求為準。
序列表
<110>廣東省林業科學研究院
<120>快速鑒定光皮梾木種群的分子檢測方法
<130>30
<170>patentinversion3.1
<210>1
<211>22
<212>dna
<213>人工序列
<400>1
catcgattaaggcagataagaa22
<210>2
<211>22
<212>dna
<213>人工序列
<400>2
tcaaatttcagagtctccacaa22
<210>3
<211>22
<212>dna
<213>人工序列
<400>3
gtggtggtgattaggaagctat22
<210>4
<211>22
<212>dna
<213>人工序列
<400>4
gtgacactggaatgaagagaga22
<210>5
<211>22
<212>dna
<213>人工序列
<400>5
aacgaaattgaaaccctagaaa22
<210>6
<211>22
<212>dna
<213>人工序列
<400>6
cagagacaaaattcaaaaagca22
<210>7
<211>22
<212>dna
<213>人工序列
<400>7
atggctatgatccaagttcttt22
<210>8
<211>22
<212>dna
<213>人工序列
<400>8
tcaaactgatcataaaacaggc22
<210>9
<211>22
<212>dna
<213>人工序列
<400>9
ggcggaagaactctatctttgt22
<210>10
<211>22
<212>dna
<213>人工序列
<400>19
tcagaacaacgtacgtacaaca22
<210>11
<211>22
<212>dna
<213>人工序列
<400>11
ggggtttatgttcttcagtagc22
<210>12
<211>22
<212>dna
<213>人工序列
<400>12
taccaccatcaatttctaaccc22
<210>13
<211>22
<212>dna
<213>人工序列
<400>13
tgtttcaagttaaggtcgattg22
<210>14
<211>22
<212>dna
<213>人工序列
<400>14
cttcaggtagaagtacccaagg22
<210>15
<211>22
<212>dna
<213>人工序列
<400>15
taaacaaaactgctgctgctat22
<210>16
<211>22
<212>dna
<213>人工序列
<400>16
agtccagaataaaaccactgct22
<210>17
<211>22
<212>dna
<213>人工序列
<400>17
caggctggtgtaactcataaaa22
<210>18
<211>22
<212>dna
<213>人工序列
<400>18
cactctcactacattgccctaa22
<210>19
<211>22
<212>dna
<213>人工序列
<400>19
agaagtattcctgtcctcatgc22
<210>20
<211>22
<212>dna
<213>人工序列
<400>20
aacccaagtaaacagaggaaac22
<210>21
<211>22
<212>dna
<213>人工序列
<400>21
ataatggctaacctacccaaga22
<210>22
<211>21
<212>dna
<213>人工序列
<400>22
cggtttgtaatagcactccac21
<210>23
<211>22
<212>dna
<213>人工序列
<400>23
ctgggacttcacacaagtagaa22
<210>24
<211>22
<212>dna
<213>人工序列
<400>24
aggtagagcattctggttccta22
<210>25
<211>21
<212>dna
<213>人工序列
<400>25
gatgatgacgattgattgagg21
<210>26
<211>22
<212>dna
<213>人工序列
<400>26
gttcagaaagaataccacccat22
<210>27
<211>23
<212>dna
<213>人工序列
<400>27
aatttgaaagaatacgtgattga23
<210>28
<211>22
<212>dna
<213>人工序列
<400>28
atgtcatttgactgtgggtaga22
<210>29
<211>22
<212>dna
<213>人工序列
<400>29
acaagcacgggcttatttatac22
<210>30
<211>22
<212>dna
<213>人工序列
<400>30
gttttcacagtgatcaaacgac22
- 還沒有人留言評論。精彩留言會獲得點贊!