用于克隆茶樹LOB基因的引物及其克隆方法和應用與流程
本發明涉及生物技術領域,尤其是茶樹lob基因、用于克隆茶樹lob基因的引物及其克隆方法和應用。
背景技術:
高等植物的頂端生長以及眾多側生器官的相繼分化和發育,是高等植物最基本的生長發育問題,直接關系到植株各部分器官的組成、個體的大小和經濟價值的高低,因而成為近年來植物功能基因組研究的一個重要熱點。
植物的莖尖分生組織(sam,shootapicalmeristem)是位于生長點的一群細胞,它從胚胎的形態發生之初開始啟動,一直維持分生狀態并貫穿植物生長發育的始終。莖尖分生組織的維持需要在中央區莖細胞和周邊區生成細胞之間保持一種平衡,這種平衡的打破往往導致側生器官原基的啟動和形成。
高等植物的側枝、葉片、花序、花器官等均為側生器官。植物的側生器官邊界區(lateralorganboundariesdomain,lbd)將葉片等側生器官(分化細胞)與頂端分生組織(干細胞)分隔開,確保器官的形成和干細胞的維持。此外,器官邊界區產成側生分生組織,進而形成側芽,影響植物株型的建成。lbd基因是最近在高等植物中發現的為植物所特有的一類新的基因,含有保守的lob(lateralorganboundaries)結構域,lob基因是植物所特有的一個基因。但由于邊界區細胞數量較少,表型不易觀察,因此對邊界區形成的正反向遺傳學研究都很困難,使得我們對邊界區形成的調控機理知之甚少。
茶葉對現代疾病如高血壓、糖尿病、輻射病、心臟病、心血管病、帕金森病、癌癥和肥胖癥等有一定的藥理功效。茶多酚是茶樹中主要的次生代謝產物,兒茶素約占茶多酚總量的70%。兒茶素生物合成主要涉及的酶類有pal、c4h、4cl、chs、chi、f3h、f3’h、f3’5’h、dfr、lar、ans和anr等,已有研究結果表明,pal、f3h、lar和dfr的表達量隨葉片成熟度增加而逐漸下降:在新芽表達量最高,新葉次之,老葉最少。
研究發現擬南芥、甜橙、亞麻薺等植物的lob基因均對植物地上部、地下部的特定器官的形成與發育具有重要影響,但對茶樹中相關lob基因的研究以及lob基因與兒茶素合成的關系的研究還沒有報道,因此對茶樹lob基因的克隆及其他分子研究尤為重要。
主要的技術文獻有:bowmanj.l.,andeshedy.,2000,formationandmaintenanceoftheshootapicalmeristem,trendsplantsci.,5(3):110-115;aida,m.,ishida,t.,tasaka,m.,1999.shootapicalmeristemandcotyledonformationduringarabidopsisembryogenesis,interactionamongthecupshapedcotyledonandshootmeristemlessgenes.development126,1563–1570;clarks.e.,2001,meristems:startyoursignaling,curr.cpin.plantbiol.,4(1):28-32;iwakawah.,uenoy.,semiartie.,onouchih.,kojimas.,tsukayah.,hasebem.,somat.,ikezakim.,machidac.,andmachiday.,2002,theasymmetricleaves2geneofarabidopsisthaliana,requiredforformationofasymmet-ricflatleaflamina,encodesamemberofanovelfamilyofproteinscharacterizedbycysteinerepeatsandaleucinezipper,plantcellphysiol.,43(5):467-478;chalfun-juniora.,frankenj.,mesj.j.,marsch-martinezn.,pereiraa.,andangenentg.c.,2005,asymmetricleaves-like1gene,amemberoftheas2/lobfamily,controlsproximal-distalpatterninginarabidopsispetals,plantmol.biol.,57(4):559-575;liuh.j.,wangs.f.,yux.b.,yuj.,hex.w.,zhangs.l.,shouh.x.,andwup.,2005,arl1,alob-domainproteinrequiredforadventitiousrootformationinrice,plantj.,43(1):47-56;abe,m.,2003.regμulationofshootepidermalcelldifferentiationbyapairofhomeodomainproteinsinarabidopsis.development130,635-643.
技術實現要素:
本發明要解決的第一個技術問題是提供一種用于克隆茶樹lob基因保守域的引物及其克隆方法,其核苷酸序列為seqidno.1,氨基酸序列為seqidno.2。為實現上述目的,本發明采用以下技術方案:
首先利用ncbi的entrez檢索系統,查找到一條擬南芥lob基因的氨基酸序列,具有seqidno.3所述的氨基酸序列。隨后利用這一序列使用blastp(通過蛋白查查找蛋白),在整個nr數據庫中查找與之相似的其它物種的氨基酸序列。
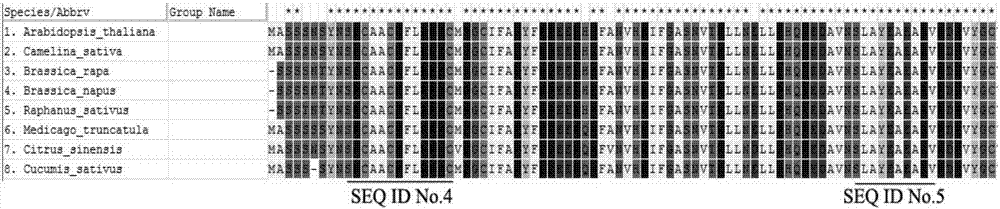
將搜索到的同一基因的不同氨基酸序列進行多序列比對,可選工具clustalw。通過序列比對,確定兩個相距47個氨基酸的保守區域,氨基酸序列分別為seqidno.4和seqidno.5所述的氨基酸序列。得到保守區域后,利用primer5.0進行簡并引物的設計。將參與多序列比對的序列中的任一條導入primer5.0中,再將其翻譯成核苷酸序列,有密碼子簡并性,其結果是有n多條彼此只相差一個核苷酸的序列群,該群可用一條有簡并性的核苷酸鏈來表示(其中r=a/g,y=c/t,m=a/c,k=g/t,s=c/g,w=a/t,h=a/c/t,b=c/g/t,v=a/c/g,d=a/g/t,n=a/c/g/t),在primer5.0中修改參數,令其在兩個距離合適的保守的nt區域內尋找引物對,要保證上下游引物都落在該簡并鏈的保守區域內。
基于上述步驟,本發明提供的克隆茶樹lob基因保守域的引物組為:
lob-degenerate-f:5’-tgygcbgcstgcaartt-3’
lob-degenerate-r:5’-gakatdgcdccdacrcagcc-3’
克隆茶樹lob基因保守域的方法包括如下步驟:提取龍井長葉rna,按照takaraprimescripttmii1ststrandcdnasynthesiskit的方法將上述rna進行逆轉錄得到cdna,以該cdna為模板,lob-degenerate-f為上游引物,lob-degenerate-r為下游引物進行pcr。獲得茶樹lob基因保守域的核苷酸序列,即seqidno.1。
本發明要解決的第二個技術問題是提供擴增茶樹lob基因5’末端、3’末端全長的特異性引物組。根據已得到的茶樹lob基因保守序列核苷酸序列,設計得到5’和3’末端擴增反應所需的引物組,其特征如下:
5’raceouterprimer:5’-catggctacatgctgacagccta-3’
5’raceinnerprimer:5’-cgcggatccacagcctactgatgatcagtcgatg-3’
cslob-5’race-gsp1:5’-aggagttgactgcgtcttcacgttggtg-3’
cslob-5’race-gsp2:5’-gagcttcgtgacgttgcttgctccaaag-3’
3’raceouterprimer:5’-taccgtcgttccactagtgattt-3’
3’raceouterprimer:5’-cgcggatcctccactagtgatttcactatagg-3’
cslob-3’race-gsp1:5’-tgcaagtttcttcgccggaaatgcatgc-3’
cslob-3’race-gsp2:5’-gagccacacaaattcgccaacgtccac-3’
本發明要解決的第三個技術問題是提供一種用于克隆茶樹lob基因全長序列的引物組及該基因的克隆方法,其特征在于用于該基因的引物分別為cslob-f(seqidno.6)和cslob-r(seqidno.7),該基因的核苷酸序列為seqidno.8。為實現上述目的,本發明采用以下技術方案:
(1)茶樹lob基因5’末端擴增反應
首先以takara5’-fullracekit提供的5’raceouterprimer和基因特異引物cslob-5’race-gsp1為引物,龍井長葉cdna為模板,進行5’末端outerpcr反應;然后再以5’raceinnerprime和基因特異引物cslob-5’race-gsp2為引物,以0.5μlouterpcr產物為模板,進行茶樹lob基因5’末端innerpcr反應。
(2)茶樹lob基因3’末端擴增反應
首先以takara3’-fullracekit提供的3’raceouterprimer和基因特異引物cslob-3’race-gsp1為引物,龍井長葉cdna為模板,進行3’末端outerpcr反應;然后再以3’raceinnerprimer和基因特異引物cslob-3’race-gsp2為引物,以0.5μlouterpcr產物為模板,進行茶樹lob基因3’末端innerpcr反應。
(3)茶樹lob基因全長cdna擴增
根據茶樹lob基因5’和3’末端測序結果分別設計了正向引物cslob-f和反向引物cslob-r,以龍井長葉cdna為模板,擴增茶樹lob基因全長序列。
本發明要解決的第四個技術問題是提供一種用于lob基因熒光定量rt-pcr的引物組,其特征在于用于定量pcr的引物分別為cslob-qrt-f(seqidno.9)和cslob-qrt-r(seqidno.10),內參基因引物分別為csgapdh-qrt-f(seqidno.11)和csgapdh-qrt-r(seqidno.12)。為實現上述目的,本發明采用以下技術方案:
提取龍井長葉不同組織(芽,第一葉,第二葉,老葉)的rna,按照takaraprimescripttmii1ststrandcdnasynthesiskit的方法將上述rna進行逆轉錄(20μl逆轉錄體系加入1μg的totalrna)得到cdna,以該cdna為模板,分別以cslob-qrt-f/cslob-qrt-r和csgapdh-qrt-f/csgapdh-qrt-r引物組,按照sybrpremixextaqii染料法熒光定量試劑盒的方法配制pcr反應液,分析不同組織中lob的表達量。
本發明的有益效果是:本發明根據已知其它物種lob基因的氨基酸及核苷酸序列設計得到擴增茶樹lob基因保守序列的引物,再根據擴增得到的茶樹lob基因保守序列,擴增茶樹lob基因的5’和3’末端序列,最終首次獲得茶樹lob基因全長序列。通過克隆茶樹lob基因的引物組和克隆方法,它能快速準確地克隆茶樹lob基因的全序列。已有研究表明,不同發育階段茶鮮葉中,新芽的兒茶素合成相關基因表達量最高,第一葉次之,成熟葉片中明顯低于第一葉。熒光定量pcr檢測結果表明,茶樹芽中lob的表達量最少,老葉中表達量相對較高,這一結果與兒茶素積累量是相反的,可能是由于lob對兒茶素的合成起負調控作用。本發明為后續茶樹lob基因的確切功能和參與的兒茶素代謝調控途徑等知識的研究奠定基礎。
附圖說明
圖1為不同植物(擬南芥、亞麻薺、蕪菁、甜橙、甘藍型油菜等)lob氨基酸序列比對圖。
圖2為cslob3’-race末端擴增的瓊脂糖凝膠電泳圖。
圖3為cslob5’-race末端擴增的瓊脂糖凝膠電泳圖。
圖4為擴增茶樹lob基因全長cdna的瓊脂糖凝膠電泳圖。
圖5為茶樹不同組織(芽、第一葉、第二葉、老葉)中lob定量pcr圖。
具體實施方式
下面結合具體實施方案對本發明做進一步的說明,這些實施例應被理解為僅為本發明的最佳實例,而非以任何方式限制本發明,凡在本發明原則之內所做的任何改進、修飾和等同替換,均應包含在本發明的范圍之內。
實施例1:龍井長葉totalrna的提取
取龍井長葉葉片組織2g于液氮中研磨,按照柱式小量植物組織抽提試劑盒說明,提取totalrna,經瓊脂糖凝膠電泳檢測質量符合要求后,凍存備用。
實施例2:茶樹lob基因保守域擴增反應
根據已知其它物種lob基因的氨基酸及核苷酸序列設計得到擴增茶樹lob基因保守序列的引物lob-degenerate-f和lob-degenerate-r,以龍井長葉cdna為模板,擴增茶樹lob基因保守域的序列。反應程序為94℃3min,94℃30s,58℃30s,72℃30s,35cycles,72℃10min。
根據已知其它物種lob基因的氨基酸及核苷酸序列設計得到擴增茶樹lob基因保守序列的引物lob-degenerate-f和lob-degenerate-r具體是:首先利用ncbi的entrez檢索系統,查找到一條擬南芥lob基因的氨基酸序列,具有seqidno.3所述的氨基酸序列。隨后利用這一序列使用blastp(通過蛋白查查找蛋白),在整個nr數據庫中查找與之相似的其它物種的氨基酸序列。
將搜索到的同一基因的不同氨基酸序列進行多序列比對,可選工具clustalw,其結果圖如圖1所示。通過序列比對,確定兩個相距47個氨基酸的保守區域,氨基酸序列分別為seqidno.4和seqidno.5所述的氨基酸序列。得到保守區域后,利用primer5.0進行簡并引物的設計。將參與多序列比對的序列中的任一條導入primer5.0中,再將其翻譯成核苷酸序列,有密碼子簡并性,其結果是有n多條彼此只相差一個核苷酸的序列群,該群可用一條有簡并性的核苷酸鏈來表示(其中r=a/g,y=c/t,m=a/c,k=g/t,s=c/g,w=a/t,h=a/c/t,b=c/g/t,v=a/c/g,d=a/g/t,n=a/c/g/t),在primer5.0中修改參數,令其在兩個距離合適的保守的nt區域內尋找引物對,要保證上下游引物都落在該簡并鏈的保守區域內。
實施例3:茶樹lob基因5’末端擴增反應
a、totalrna去磷酸化處理
按照takara5’-fullracekit說明,使用alkalinephosphatase(ciap)對totalrna中裸露的5’磷酸基團進行去磷酸反應。去磷酸反應液體系為茶樹totalrna(1μg/μl)1μl,rnaseinhibitor(40u/μl)1μl,10×alkalinephosphatasebuffer(mgcl2free)5μl,alkalinephosphatase(calfintestine)(16u/μl)0.6μl,rnasefreedh2o46.9μl。50℃反應1小時后。向上述反應液中加入20μl的3mch3coona(ph5.2),130μl的rnasefreedh2o,充分混勻。再加入200μl的苯酚/氯仿/異戊醇(25:24:1),充分混勻后13,000×g室溫離心5min,將上層水相轉移至新的microtube中。再加入200μl的氯仿,充分混勻后13,000×g室溫離心5min,將上層水相轉移至新的microtube中。加入2μl的nacarrier均勻混合后,加入200μl的異丙醇,冰上冷卻10min,13,000×g4℃離心20min,棄上清,加入500μl70%的乙醇漂洗,13,000×g4℃離心5min,棄上清后干燥,最后加入7μl的rnasefreedh2o溶解沉淀,得到ciap-treatedrna。
b、mrna“去帽子”反應。
使用tobaccoacidpyrophosphatase(tap)去掉mrna的5’帽子結構,保留一個磷酸基團。反應體系為ciap-treatedrna7μl,rnaseinhibitor(40u/μl)1μl,10×tapreactionbuffer1μl,tobaccoacidpyrophosphatase(0.5u/μl)1μl,37℃反應1小時,得到ciap/tap-treatedrna。
c、5’raceadaptor的連接
首先配置下列溶液:ciap/tap-treatedrna5μl,5’raceadaptor(15μm)1μl,rnasefreedh2o4μl。上述溶液65℃反應1小時后,加入20μl的3mch3coona(ph5.2),140μl的rnasefreedh2o,充分混勻。再加入200μl的苯酚/氯仿/異戊醇(25:24:1),充分混勻后13,000×g室溫離心5min,將上層水相轉移至新的microtube中。再加入200μl的氯仿,充分混勻后13,000×g室溫離心5min,將上層水相轉移至新的microtube中。加入2μl的nacarrier均勻混合后,加入200μl的異丙醇,冰上冷卻10min,13,000×g4℃離心20min,棄上清,加入500μl70%的乙醇漂洗,13,000×g4℃離心5min,棄上清后干燥,最后加入6μl的rnasefreedh2o溶解沉淀,得到ligatedrna。
d、反轉錄反應。
反應體系為ligatedrna6μl,random9mers(50μm)0.5μl,dntp(10mmeach)1μl,rnaseinhibitor(40u/μl)0.25μl,reversetranscriptasem-mlv(rnaseh-)(200u/μl)0.25μl。反應條件為30℃10min,42℃1hr,72℃15min,即可得到用于擴增5’末端的龍井長葉cdna。
e、outerpcr反應。
以takara5’-fullracekit提供的5’raceouterprimer和基因特異引物cslob-5’race-gsp1為引物,龍井長葉cdna為模板,進行5’末端outerpcr反應;擴增體系為龍井長葉cdna2μl,1×cdnadilutionbufferii8μl,10×lapcrbuffer(mg2+free)4μl,mgcl2(25mm)3μl,takaralataq(5u/μl)0.25μl,cslob-5’race-gsp1(10μm)2μl,5’raceouterprimer(10μm)2μl,dh2o28.75μl。pcr反應條件為94℃3min,94℃30s,55℃30s,72℃1min,20cycles,72℃10min。
f、innerpcr反應。
以5’raceinnerprimer和基因特異引物cslob-5’race-gsp2為引物,以1μlouterpcr產物為模板,進行茶樹lob基因5’末端innerpcr反應。反應體系和擴增程序同outerpcr反應。pcr產物經電泳檢測,結果如圖2所示。
實施例4:茶樹lob基因3’末端擴增反應
a、反轉錄反應。
反應體系為totalrna(500ng/μl)1μl,3’raceadaptor(5μm)1μl,5×primescriptbuffer2μl,dntpmixture(10mmeach)1μl,rnaseinhibitor(40u/μl)0.25μl,primescriptrtase(200u/μl0.25μl)0.25μl,rnasefreedh2o4.5μl。反應條件42℃60min,70℃15min,反應結束即得到用于擴增3’末端的龍井長葉cdna。
b、outerpcr反應。
以takara3’-fullracekit提供的3’raceouterprimer和基因特異引物cslob-3’race-gsp1為引物,龍井長葉cdna為模板,進行3’末端outerpcr反應;擴增體系為龍井長葉cdna2μl,1×cdnadilutionbufferii8μl,10×lapcrbuffer(mg2+free)4μl,mgcl2(25mm)3μl,takaralataq(5u/μl)0.25μl,cslob-3’race-gsp1(10μm)2μl,3’raceouterprimer(10μm)2μl,dh2o28.75μl。pcr反應條件為94℃3min,94℃30s,55℃30s,72℃1min,20cycles,72℃10min。
c、innerpcr反應。
以3’raceinnerprimer和基因特異引物cslob-3’race-gsp2為引物,以1μlouterpcr產物為模板,進行茶樹lob基因3’末端innerpcr反應。反應體系和擴增程序同outerpcr反應。pcr產物經電泳檢測,結果如圖3所示。
實施例5:茶樹lob基因全長cdna序列擴增。
根據茶樹lob基因5’和3’末端測序結果分別設計了正向引物cslob-f和反向引物cslob-r,以龍井長葉cdna為模板,擴增茶樹lob基因全長序列。反應程序為94℃3min,94℃30s,58℃30s,72℃30s,35cycles,72℃10min。pcr產物電泳結果如圖4所示。
實施例6:pcr產物的克隆純化與篩選。
pcr產物經1.5%瓊脂糖凝膠電泳后,用膠回收試劑盒對pcr產物進行回收,經純化后與takara公司t載體pmd18-tvector進行連接,用熱激法導入大腸桿菌dh5α感受態中并在氨芐青霉素平板上篩選陽性克隆。分子檢測確定后的陽性克隆子送杭州擎科生物公司完成測序。
實施例7:熒光定量pcr檢測茶樹不同組織中lob的表達量
提取龍井長葉不同組織(芽,第一葉,第二葉,老葉)的rna,按照takaraprimescripttmii1ststrandcdnasynthesiskit的方法將上述rna進行逆轉錄(20μl逆轉錄體系加入1μg的totalrna)得到cdna,以該cdna為模板,利用本發明所述的引物cslob-qrt-f、cslob-qrt-r、csgapdh-qrt-f和csgapdh-qrt-r為引物,按照下列組分配制pcr反應液:
pcr擴增程序如下:94℃3min,94℃20s,58℃20s,72℃20s,40cycles,72℃收集熒光,結果如圖5所示。
seqidno.1:
tgcgcggcgtgcaagtttcttcgccggaaatgcatgccgggatgcatattcgcgccatattttccaccggaggagccacacaaattcgccaacgtccacaaaatctttggagcaagcaacgtcacgaagctcctcaacgagctcctccctcaccaacgtgaagacgcagtcaactccttagcctatgaggccgaggcacgtgtccgtgaccccgtctatggctgcgtaggcgccatctc
seqidno.2:
caackflrrkcmpgcifapyfppeephkfanvhkifgasnvtkllnellphqredavnslayeaearvrdpvygcvgai
seqidno.3:
masssnsynspcaackflrrkcmpgcifapyfppeephkfanvhkifgasnvtkllnellphqredavnslayeaearvrdpvygcvgaisylqrqvhrlqkeldaanadlahyglstsaagapgnvvdlvfqpqplpsqqlpplnpvyrlsgaspvmnqmprgtggsygtflpwnnghdqqggnm
seqidno.4:spcaackflrrkc
seqidno.5:slayeaearv
seqidno.6:atggcgtcgtcatcaaactcatacaac
seqidno.7:tcaaatatttcctccagaagggctattg
seqidno.8:
atggcgtcgtcatcaaactcatacaactcaacatgcgcggcgtgcaagtttcttcgccggaaatgcatgccgggatgcatattcgcgccatattttccaccggaggagccacacaaattcgccaacgtccacaaaatcttcggcgcaagcaacataagtaagctcctcaatgaaatcctccctcaccaaagagatgatgcagtgagttccctcgcctacgaagccgaagcacgagtgcgagatccagtgtacggttgtgttggtgctatctccttcctccaaagacaggttgagaagctccagaaagatctagaagtcgccaatgccaatttgattcactga
seqidno.9:5’-gaaatcctccctcaccaaagag-3’
seqidno.10:5’-aggagatagcaccaacacaac-3’
seqidno.11:5’-ttggcatcgttgagggtct-3’
seqidno.12:5’-cagtgggaacacggaaagc-3’
sequencelisting
<110>浙江省農業科學院
<120>用于克隆茶樹lob基因的引物及其克隆方法
<130>1
<160>12
<170>patentinversion3.3
<210>1
<211>239
<212>dna
<213>人工合成
<400>1
tgcgcggcgtgcaagtttcttcgccggaaatgcatgccgggatgcatattcgcgccatat60
tttccaccggaggagccacacaaattcgccaacgtccacaaaatctttggagcaagcaac120
gtcacgaagctcctcaacgagctcctccctcaccaacgtgaagacgcagtcaactcctta180
gcctatgaggccgaggcacgtgtccgtgaccccgtctatggctgcgtaggcgccatctc239
<210>2
<211>79
<212>prt
<213>人工合成
<400>2
cysalaalacyslyspheleuargarglyscysmetproglycysile
151015
phealaprotyrpheproproglugluprohislysphealaasnval
202530
hislysilepheglyalaserasnvalthrlysleuleuasngluleu
354045
leuprohisglnarggluaspalavalasnserleualatyrgluala
505560
glualaargvalargaspprovaltyrglycysvalglyalaile
657075
<210>3
<211>186
<212>prt
<213>人工合成
<400>3
metalaserserserasnsertyrasnserprocysalaalacyslys
151015
pheleuargarglyscysmetproglycysilephealaprotyrphe
202530
proproglugluprohislysphealaasnvalhislysilephegly
354045
alaserasnvalthrlysleuleuasngluleuleuprohisglnarg
505560
gluaspalavalasnserleualatyrglualaglualaargvalarg
65707580
aspprovaltyrglycysvalglyalailesertyrleuglnarggln
859095
valhisargleuglnlysgluleuaspalaalaasnalaaspleuala
100105110
histyrglyleuserthrseralaalaglyalaproglyasnvalval
115120125
aspleuvalpheglnproglnproleuproserglnglnleupropro
130135140
leuasnprovaltyrargleuserglyalaserprovalmetasngln
145150155160
metproargglythrglyglysertyrglythrpheleuprotrpasn
165170175
asnglyhisaspglnglnglyglyasnmet
180185
<210>4
<211>13
<212>prt
<213>人工合成
<400>4
serprocysalaalacyslyspheleuargarglyscys
1510
<210>5
<211>10
<212>prt
<213>人工合成
<400>5
serleualatyrglualaglualaargval
1510
<210>6
<211>27
<212>dna
<213>人工合成
<400>6
atggcgtcgtcatcaaactcatacaac27
<210>7
<211>28
<212>dna
<213>人工合成
<400>7
tcaaatatttcctccagaagggctattg28
<210>8
<211>342
<212>dna
<213>人工合成
<400>8
atggcgtcgtcatcaaactcatacaactcaacatgcgcggcgtgcaagtttcttcgccgg60
aaatgcatgccgggatgcatattcgcgccatattttccaccggaggagccacacaaattc120
gccaacgtccacaaaatcttcggcgcaagcaacataagtaagctcctcaatgaaatcctc180
cctcaccaaagagatgatgcagtgagttccctcgcctacgaagccgaagcacgagtgcga240
gatccagtgtacggttgtgttggtgctatctccttcctccaaagacaggttgagaagctc300
cagaaagatctagaagtcgccaatgccaatttgattcactga342
<210>9
<211>22
<212>dna
<213>人工合成
<400>9
gaaatcctccctcaccaaagag22
<210>10
<211>21
<212>dna
<213>人工合成
<400>10
aggagatagcaccaacacaac21
<210>11
<211>19
<212>dna
<213>人工合成
<400>11
ttggcatcgttgagggtct19
<210>12
<211>19
<212>dna
<213>人工合成
<400>12
cagtgggaacacggaaagc19
- 還沒有人留言評論。精彩留言會獲得點贊!
- 一組小熊貓基因的微衛星引物的制作方法與工藝
- 羊肚菌交配型決定基因鑒定專用引物及出菇能力預測方法與流程
- 一種KRAS基因的檢測引物組、其構成的反應體系及應用的制作方法與工藝
- MTHFR?A1298C基因多態性快速檢測引物、分子信標、試劑盒及檢測方法與流程
- 用于檢測人類VKORC1與CYP2C9基因多態性的引物、探針、試劑盒及方法與流程
- 一種牛卷毛基因快速篩選的引物、方法及試劑盒與流程
- KCNQ1OT1基因、aHIF基因及其引物的應用以及用于診斷冠心病的試劑盒的制作方法與工藝
- 用于檢測人類ALDH2基因多態性的引物、探針、試劑盒及方法與流程
- 一種檢測耳聾基因的引物及其應用的制造方法與工藝
- 海島棉跨膜蛋白基因、引物及其應用的制造方法與工藝
- 玉米低鎂條件下特異性轉運鎂離子基因ZmMGT10及其應用的制造方法與工藝
- CRISPR?Cas9特異性敲除豬SLA?3基因的方法及用于特異性靶向SLA?3基因的sgRNA與制造工藝
- 毛酸漿的分子特異性標記引物及鑒別方法與制造工藝
- 一種檢測CYP2C9基因多態性的ARMS引物的制造方法與工藝
- 構建在海馬體區域特異性敲除IKKα基因的小鼠模型的方法及打靶載體和試劑盒與制造工藝
- 使用重疊非等位基因特異性引物和等位基因特異性阻斷劑寡核苷酸的組合物進行等位基因特異性擴增的制造方法與工藝
- 一種利用線粒體D-loop區特異區域鑒定黿的標記引物及方法
- 一種用于檢測大豆擬莖點種腐病菌的特異性lamp引物組合物及其應用
- 一種針對膨脹污泥中Eikelboom Type 0092型絲狀菌群特異性引物和方法
- 脯氨酸特異性蛋白酶基因及其應用