一種構建穩定過表達CXCR4的iPSCs的方法及其應用與流程
本發明屬于醫藥技術領域,具體涉及一種構建穩定過表達cxcr4的ipscs的方法及其應用。
背景技術:
具備多向分化能力的誘導性多能干細胞(inducedpluripotentstemcells,ipscs)是治療心肌梗死的理想種子細胞,但心梗后局部微環境的變化以及較低細胞移植歸巢率限制了其應用。
基質衍生因子(stromalderivedfactor-1,sdf-1)即cxcl12,可在心肌梗死后的心梗區域局部上調,cxcl12通過與其獨特受體cxcr4相互作用可促進胚胎干細胞遷移、存活,在胚胎生長發育及器官形成中發揮著極重要作用。
tale(transcriptionactivator-like(tal)effector)靶向基因操作技術是一項嶄新的分子生物學工具,是基因功能研究領域的一項重大技術突破。研究人員發現,來自植物病原菌xanthomonas的tal蛋白的核酸結合域的氨基酸序列與其靶位點的核酸序列有恒定的對應關系。基于這一對應關系,利用tal的序列模塊構建特異性結合任意dna序列的重組蛋白,可實現操作目標基因的目的。
目前尚無通過talens技術構建穩定過表達cxcr4-ipscs以提高ipscs在治療心肌梗死方面的研究。
技術實現要素:
為了克服現有技術的缺點與不足,本發明的首要目的在于提供一種構建穩定過表達cxcr4的ipscs的方法。
本發明研究發現,talens技術可穩定構建穩定過表達cxcr4-ipscs,且不改變其分化潛能及其基本形態,體外實驗證實可有效提高其細胞歸巢率。因此,基于talens技術構建的cxcr4-ipscs可提高干細胞歸巢效率。
本發明的另一目的在于提供通過上述構建方法構建得到的穩定過表達cxcr4的ipscs。
本發明的再一目的在于提供上述穩定過表達cxcr4的ipscs的應用。
本發明的目的通過下述技術方案實現:
一種構建穩定過表達cxcr4的ipscs的方法,包括如下步驟:
利用tales技術誘導cxcr4基因啟動子區域去甲基化,通過cxcr4慢病毒顆粒轉染ipscs,得到穩定過表達cxcr4的ipscs,從而實現靶向基因的過表達。
一種構建穩定過表達cxcr4的ipscs的方法,所述的talens技術:
具體包括如下步驟:
1)cxcr4基因(nm_022205)的啟動子區富含cpg甲基化位點,我們已經成功合成了能夠識別并定位cxcr4基因啟動子區dna序列的tale基因,這兩個人工合成基因分別定位cxcr4a:5′-tagacccacactgacttaaaac-3′和cxcr4b:5′-ttcaattttgttgcctggtgca-3′。
2)然后將這兩個基因分別同dna去甲基化酶tet1的催化域(catalyticdomain)融合,并在tet1c的末端用t2a技術連接綠色熒光蛋白(gfp),構建cxcr4a-tet1c-gfp和cxcr4b-tet1c-gfp質粒;最后將這兩個融合基因分別克隆到慢病毒質粒pcdhcd527a。
3)慢病毒的包裝和濃縮:將慢病毒包裝質粒和載體質粒cxcr4-tet1c-gfp以1:1的摩爾比例混合,在lipofectamine2000的作用下共轉染293ft細胞,置于37℃、體積分數5%co2的培養箱中培養;12h后換液,加入新鮮293ft細胞培養液;72h后觀察有強綠色熒光及細胞融合現象時收集培養上清,4℃、4500r/min離心15min,將病毒上清液用0.45μm濾膜過濾以去除細胞碎片,4℃、50000×g高速離心90min;用rnase-freel-dmem懸浮病毒顆粒沉淀,-80℃凍存備用;同法構建只含綠色熒光蛋白的空病毒;
4)cxcr4-tet1c-gfp病毒顆粒穩定感染ipscs:將對數生長期ipscs消化并離心后,以分化培養基重懸成濃度1×106/l細胞懸液,將病毒cxcr4-tet1c-gfp和空病毒以10:1轉染誘導性多能干細胞的;每天更換1次培養基;病毒感染24~48h后,更換新鮮培養液;并同時使用氨芐青霉素進行篩選;得到穩定過表達cxcr4的ipscs。
一種穩定過表達cxcr4的ipscs,通過上述方法構建得到。
所述的穩定過表達cxcr4的ipscs在增強細胞遷移特性中的應用。
本實驗原理:本申請通過轉錄激活因子樣效應物核酸酶(transcriptionactivator-likeeffectornucleases,talens)技術構建穩定表達cxcr4的誘導性多能干細胞(inducedpluripotentstemcells,ipscs),ipscs在修復急性心肌梗死后心肌上具有巨大臨床應用前景,但其細胞移植歸巢效率低下及梗死部位惡劣的微環境造成其治療效果受限,通過生物手段提高干細胞歸巢率及治療效率在預防和治療心肌損傷相關心血管疾病(如缺血性心臟病、動脈粥樣硬化、高血壓)等方面有廣闊的應用前景。
本發明通過cxcr4慢病毒顆粒轉染ipscs實現cxcr4過表達,流式細胞儀技術檢驗慢病毒顆粒對ipscs凋亡率影響,bsp(bisulfitesequencingpcr)、倒置顯微鏡、共聚焦顯微鏡和westernblots證實cxcr4可穩定在細胞表面表達。體外遷移實驗證實可顯著提高其細胞遷移率。
本發明相對于現有技術,具有如下的優點及效果:
本發明中通過tales技術構建穩定表達cxcr4的ipscs,并通過qpcr、形態學鑒定和bsp證實過表達cxcr4后對ipscs的干性維持基因無影響,且可顯著提高體外細胞遷移率。
附圖說明
圖1是定位cxcr4a和cxcr4b的示意圖。
圖2是cxcr4b-tet1c-gfp質粒的構建示意圖。
圖3是慢病毒質粒pcdhcd527a的骨架系統。
圖4是ipscs中cxcr4表達情況,其中,a:共聚焦顯微鏡下cxcr4表達情況(600×);b:倒置顯微鏡下的ipscs轉染前后的形態學改變(40×);c:mrna(cxcr4、oct4、c-myc)表達情況;d:cxcr4蛋白表達情況;e:cxcr4蛋白表達的相對定量(control:對照組,細胞不做任何處理;cxcr4-ipscs:cxcr4過表達的ipscs;且cxcr4代表cxcr4b)。
圖5是cxcr4慢病毒顆粒對ipscs凋亡影響;其中,a:流式細胞圖;b:凋亡數據分析(control:對照組,細胞不做任何處理;cxcr4-ipscs:cxcr4過表達的ipscs;emptyvectoripscs:空病毒轉染組;且cxcr4代表cxcr4b)。
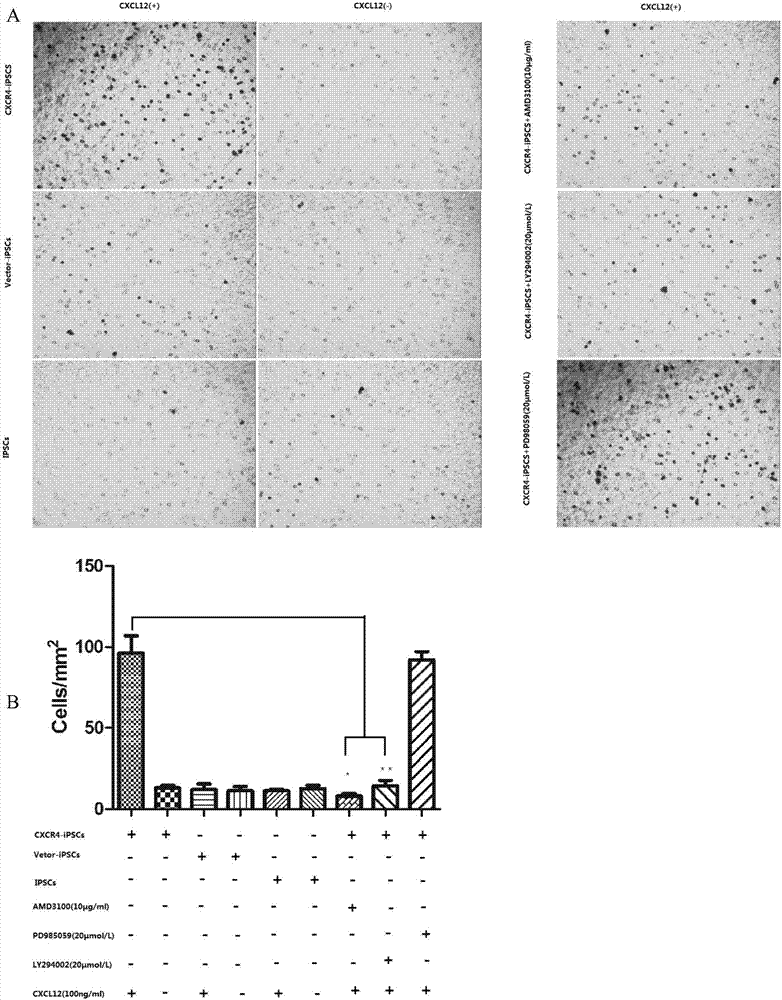
圖6是體外細胞遷移實驗;其中,a:transwell形態圖;b:細胞遷移率柱狀圖(cxcr4代表cxcr4b)。
圖7是cxcr4、oct4、c-myc甲基化狀態(cxcr4代表cxcr4b)。
具體實施方式
下面結合實施例及附圖對本發明作進一步詳細的描述,但本發明的實施方式不限于此。
下列實施例中未注明具體實驗條件的實驗方法,通常按照常規實驗條件或按照制造廠商所建議的實驗條件。未注明具體來源的試劑及生物材料,均為市售產品。
實施例1制備ipscs
(1)滋養層(mef)的制備:0.1%(體積分數)的明膠加入t25培養瓶,搖勻覆蓋即可,于37℃細胞培養箱靜置20min后將其吸除,加入5~6ml預熱37℃的mef培養液,與此同時將小鼠胚胎成纖維細胞(mef)(購自上海中科院細胞庫)從液氮中快速取出,置于37℃水浴中快速融解并立即用體積分數為75%的酒精擦拭凍存管后拿到超凈臺內,將凍存管內的細胞懸液轉移至含mef培養液的15ml離心管內,以1000rpm離心5min,棄上清液重懸后加入到t25培養瓶中,放置到co2恒溫培養箱中,培養24h后得到滋養層即可加入ipscs;
(2)ipscs的培養以及傳代:步驟(1)中培養24h后得到的滋養層,4倍顯微鏡下可見已經均勻鋪滿t25培養瓶,此時可迅速從液氮中取出ipscs(中科院上海生科院生化細胞所和中科院上海生科院生化細胞所干細胞平臺提供)凍存管,復蘇過程類似小鼠胚胎成纖維細胞,將復蘇的ipscs直接傳入mef鋪板的t25培養瓶,置于37℃細胞培養箱,每日換液并且觀察細胞集落形態的變化。待細胞集落長到合適大小及時傳代,pbs(不含鈣鎂離子)輕輕沖洗一遍后加入0.25%(體積分數)胰酶(含edta)消化,小鼠ipscs培養液終止消化,以1000rpm離心5min,棄上清加培養液吹打制備成單細胞懸液。
實施例2制備cxcr4慢病毒顆粒
一種構建穩定過表達cxcr4的ipscs的方法,所述的talens技術:
基本方法如下:
1)cxcr4基因(nm_022205)的啟動子區富含cpg甲基化位點,我們已經成功合成了能夠識別并定位cxcr4基因啟動子區dna序列的tale基因,這兩個人工合成基因分別定位cxcr4a:5′-tagacccacactgacttaaaac-3′和cxcr4b:5′-ttcaattttgttgcctggtgca-3′(如圖1所示)。
2)然后將這兩個基因分別同dna去甲基化酶tet1的催化域(catalyticdomain)融合,并在tet1c的末端用t2a技術連接綠色熒光蛋白(gfp),構建cxcr4a-tet1c-gfp和cxcr4b-tet1c-gfp質粒(如圖2所示,cxcr4代表cxcr4b)。最后將這兩個融合基因分別克隆到慢病毒質粒pcdhcd527a(骨架系統見圖3所示)。
3)慢病毒的包裝和濃縮:將慢病毒包裝質粒和載體質粒cxcr4-tet1c-gfp以1:1的摩爾比例混合,在lipofectamine2000的作用下共轉染293ft細胞,置于37℃、體積分數5%co2的培養箱中培養。12h后換液,加入新鮮293ft細胞培養液。72h后觀察有強綠色熒光及細胞融合現象時收集培養上清,4℃、4500r/min離心15min,將病毒上清液用0.45μm濾膜過濾以去除細胞碎片,4℃、50000×g高速離心90min。用rnase-freel-dmem懸浮病毒顆粒沉淀,-80℃凍存備用。同法構建只含增強型綠色熒光蛋白的空病毒。
4)慢病毒的滴度測定:接種293ft細胞于6孔板,每孔接種2×105個細胞,37℃培養過夜;第2天將慢病毒溶液用無血清dmem(不含抗生素)按1×10-2,1×10-3,1×10-4,1×10-5,1×10-6,1×10-7進行系列稀釋,將其分別加入到相應的孔中,加入聚酰胺(polybrene)終濃度至8mg/l,置37℃、體積分數5%co2培養箱培養。12h后全量換液,加入新鮮的293ft細胞培養液。第4天觀察熒光表達情況,熒光細胞數隨稀釋倍數增加而減少,計數出表達熒光的細胞個數,將得到的數值乘以相應的稀釋倍數就得到病毒原液的滴度數。
5)cxcr4-tet1c-gfp病毒顆粒穩定感染ipscs:將對數生長期ipscs消化并離心后,以分化培養基重懸成濃度1×106/l細胞懸液,將病毒cxcr4-tet1c-gfp和空病毒以10:1轉染誘導性多能干細胞的。每天更換1次培養基。病毒感染24~48h后,更換新鮮培養液。并同時使用氨芐青霉素進行篩選。
所述的分化培養基為含有體積分數分別為1%非必需氨基酸、1%谷氨酰胺、0.25%β-巰基乙醇,15%胎牛血清的dmem。
6)流式細胞儀技術:制備單細胞細胞懸液后,冰預冷的70%的乙醇固定,4℃,1~2小時,細胞重懸,400目的篩網過濾1次,500~1000r/min離心5min,棄去pbs,1mlpi染液染色,4℃避光30min,pi用氬離子激發熒光,激光光波波長為488nm,發射光波波長大于630nm,產生紅色熒光分析pi熒光強度的直方圖也可分析前散射光對側散射光的散點圖。
cxcr4慢病毒顆粒對ipscs凋亡影響,如圖5所示(control:對照組,細胞不做任何處理;cxcr4-ipscs:cxcr4過表達的ipscs;emptyvectoripscs:空病毒轉染組;且cxcr4代表cxcr4b);其中,a:流式細胞圖;b:凋亡數據分析。結果表明:相比對照組而言,cxcr4慢病毒和空病毒對ipscs凋亡均存在一定的影響,可引起有效的凋亡;但cxcr4慢病毒和空病毒兩者間凋亡和增殖無顯著差異。
實施例3cxcr4過表達情況
單細胞克隆株在擴增消化離心后進行細胞爬片,pbs緩沖溶液沖洗3次后,小牛血清37℃孵育30min,一抗4℃過夜,二抗37℃孵育30min,dapa染色3~5min,共聚焦顯微鏡下觀察。
單細胞懸液經過pbs緩沖液沖洗后,實時熒光定量pcr(qpcr)技術:各組ipscs經預處理48h后,按trizol操作說明書抽提樣品細胞的總rna。紫外分光光度計定量后,取總rna用反轉錄酶逆轉錄得到cdna;于pcr儀上進行擴增,反應程序為:95℃首輪變性1min(stage1);95℃變性15s,60℃退火和延伸45s(stage2),40個循環后;95℃15s,60℃15s(stage3,dissociationstage)。為排除樣品上樣量差異造成的影響,每個基因的ct值都用管家基因gapdh的ct值進行校正。
ipscs中cxcr4表達情況,如圖4所示(control:對照組,細胞不做任何處理;cxcr4-ipscs:cxcr4過表達的ipscs;且cxcr4代表cxcr4b);其中,a:共聚焦顯微鏡下cxcr4表達情況(600×);b:倒置顯微鏡下的ipscs轉染前后的形態學改變(40×);c:mrna(cxcr4、oct4、c-myc)表達情況;d:cxcr4蛋白表達情況;e:cxcr4蛋白表達的相對定量。結果表明:共聚焦顯微鏡下可見cxcr4可在ipscs上大量表達,且倒置顯微鏡下無顯著形態學改變;qpcr技術結果提示cxcr4mrna水平相比對照組增加了1.8倍(p<0.05),且對分化潛能控制基因oct-4和c-myc的改變無統計學意義(p>0.05)。westernblot結果相比對照組而言提示ipscs經過talens的修飾cxcr4蛋白顯著提高6倍(p<0.05)。
實施例4cxcr4對ipscs遷移的影響
1)選用5mm孔徑的transwell小室(bectondickinsonlabware,usa);
2)ipscs胰酶消化后重懸,細胞計數板計數;
3)實驗分組:
①cxcr4-ipscs組(cxcr4過表達的ipscs);
②ipscs組(ipscs不做任何處理);
③vector-ipscs組(空病毒轉染組);
④cxcr4-ipscs+cxcl12組;
⑤ipscs+cxcl12組;
⑥vector-ipscs+cxcl12組;
⑦amd3100+cxcr4-ipscs+cxcl12組;
⑧pd98059+cxcr4-ipscs+cxcl12組組;
⑨ly294002+cxcr4-ipscs+cxcl12組;
選用8mm孔徑的transwell小室,上室按實驗分組分別加入5×104/孔的細胞,以及cxcr4特異性阻斷劑amd3100(10μg/ml)、erk1/2特異阻斷劑pd98059(20μmol/l)、akt特異阻斷劑ly294002(20μmol/l)預處理1h,下室分別加入含或不含cxcl12(100ng/ml)的無血清培養基;37℃、co2培養箱中孵育4小時,棄培養液,pbs沖洗2次,用棉簽刮輕輕刮除聚碳酸酯膜上層的細胞,將transwell小室室溫下置于4%多聚甲醛中固定10min,pbs沖洗2-3次,1%結晶紫室溫下染色15min;pbs沖洗2~3次,倒置相差顯微鏡下隨機挑選5個視野計數,重復實驗三次計數統計。
體外細胞遷移實驗;結果如圖6所示(cxcr4代表cxcr4b);其中,a:transwell形態圖;b:細胞遷移率數據分析。結果表明:transwell下室均未放置100ng/mlcxcl12的ipscs組、cxcr4-ipscs組、vector-ipscs組各組間細胞遷移數的差異無統計學意義(p>0.05)。100ng/mlcxcl12預處理4h后,相比ipscs+cxcl12組的細胞遷移數而言,實驗組cxcr4-ipscs+cxcl12組遷移至小室下層的細胞數量具有顯著的統計學意義(p<0.05),而vector-ipscs組的遷移細胞數無統計學意義(p>0.05);經過cxcr4受體拮抗劑amd3100和ly294002預處理1h后,相比cxcr4-ipscs+cxcl12組的高效遷amd3100+cxcr4-ipscs+cxcl12組的細胞遷移顯著減少(p<0.05),但pd98059對cxcr4-ipscs的遷移無明顯的影響(p>0.05)。
實施例5ipscs過表達cxcr4后對分化潛能基因oct-4和c-myc的影響
利用qpcr技術(同上)以及bsp技術證實該方法可使ipscs穩定cxcr4的同時,并不影響分化潛能基因。bsp技術:利用promegawizardgenomicdnapurificationkit(貨號cat.#a1125)抽提dna,qiagen公司的epitectbisulfitekit(貨號cat.no.59104)亞硫酸氫鹽修飾,再進行基因的克隆測序,bsp擴增的甲基化的引物:
bsp-cxcr4:183bp,15cpg,
bsp-cxcr4-f:5′-ggttttggatttatattgatttaaaatata-3′,
bsp-cxcr4-r:5′-ccaaacaacaaaattaaaatttctaac-3′;
bsp-c-myc:232bp,16cpg,
bsp-c-myc-f:5′-tttgttttttgaagggtagggt-3′,
bsp-c-myc-r:5′-aaaacaaaacccctctcactc-3′;
bsp-oct4:232bp,15cpg,
bsp-oct4-f:5′-tggttgagtgggttgtaaggataggt-3′,
bsp-oct4-r:5′-actccaaccctactaacccatcacc-3′。
cxcr4、oct4、c-myc甲基化狀態,如圖7所示(cxcr4代表cxcr4b)。結果表明:將經過慢病毒顆粒處理篩選以及未處理的ipscs送至廣州艾基生物公司測序,可見cxcr4-ipscs上cxcr4的cpg位點有效去甲基化激活cxcr4內源性表達,而對照組以甲基化為主(p<0.05)。oct4和c-myc基因的甲基化和去甲基化無顯著的改變(p>0.05),cxcr4的表達量與其去甲基化狀態成正相關關系。
上述實施例為本發明較佳的實施方式,但本發明的實施方式并不受上述實施例的限制,其他的任何未背離本發明的精神實質與原理下所作的改變、修飾、替代、組合、簡化,均應為等效的置換方式,都包含在本發明的保護范圍之內。
sequencelisting
<110>暨南大學
<120>一種構建穩定過表達cxcr4的ipscs的方法及其應用
<130>1
<160>8
<170>patentinversion3.5
<210>1
<211>22
<212>dna
<213>artificialsequence
<220>
<223>cxcr4a
<400>1
tagacccacactgacttaaaac22
<210>2
<211>22
<212>dna
<213>artificialsequence
<220>
<223>cxcr4b
<400>2
ttcaattttgttgcctggtgca22
<210>3
<211>30
<212>dna
<213>artificialsequence
<220>
<223>bsp-cxcr4-f
<400>3
ggttttggatttatattgatttaaaatata30
<210>4
<211>27
<212>dna
<213>artificialsequence
<220>
<223>bsp-cxcr4-r
<400>4
ccaaacaacaaaattaaaatttctaac27
<210>5
<211>22
<212>dna
<213>artificialsequence
<220>
<223>bsp-c-myc-f
<400>5
tttgttttttgaagggtagggt22
<210>6
<211>21
<212>dna
<213>artificialsequence
<220>
<223>bsp-c-myc-r
<400>6
aaaacaaaacccctctcactc21
<210>7
<211>26
<212>dna
<213>artificialsequence
<220>
<223>bsp-oct4-f
<400>7
tggttgagtgggttgtaaggataggt26
<210>8
<211>25
<212>dna
<213>artificialsequence
<220>
<223>bsp-oct4-r
<400>8
actccaaccctactaacccatcacc25
- 還沒有人留言評論。精彩留言會獲得點贊!