一種濾除DNase高通量測序數據中DNA堿基傾向性偏差的方法與流程
本發明屬于分子生物信息檢測與分析領域,具體涉及一種有效提高DNase高通量測序數據的檢測信息準確性的濾除DNase高通量測序數據中DNA堿基傾向性偏差的方法。
背景技術:
目前,DNA蛋白結合位點的檢測主要采用染色質免疫共沉淀技術(Chromatin Immunoprecipitation,ChIP)。而將ChIP實驗結果與高通量測序技術相結合的ChIP-Seq技術,則能有效地在全基因組范圍內檢測目的功能蛋白在DNA上的結合位點。ChIP-Seq的原理是:首先通過染色質免疫共沉淀技術(ChIP)利用與目的蛋白特異性結合的酶來富集結合有目的蛋白的DNA片段,并對其進行純化與文庫構建。然后對富集得到的DNA片段進行高通量測序,再將測序獲得的數百萬條讀數序列精確定位到基因組上,從而獲得全基因組范圍內結合有目的蛋白的DNA區段信息,進而通過各種分析算法得到目的蛋白DNA結合位點。
然而,ChIP-Seq技術也有諸多不足之處,首先是富集目的蛋白的結合酶具有特異性,從而導致某些蛋白因找不到合適的特異結合酶而無法進行檢測;其次,一次實驗只能檢測一種蛋白,耗時耗力,成本高,無法大規模使用;第三,更為重要的是,由于實驗獲取的與目的蛋白結合的DNA片段較長,測序時只能對其兩端進行部分測序,由于測序區域并不是結合位點本身,因此,ChIP-Seq技術對DNA蛋白結合位點的檢測分辨率無法達到單堿基。
針對上述問題,近幾年產生了一種新的DNA蛋白結合位點檢測技術--基于DNase高通測序信息的DNA蛋白結合位點檢測技術,即DNase-Seq技術。DNase-Seq的原理是:首先利用DNase核酸剪切酶對DNA進行酶切處理。則沒有DNA蛋白結合的DNA區域將被DNase核酸剪切酶隨機地切斷,而有DNA蛋白結合的DNA區域由于受到結合蛋白的阻礙特異性不被切斷。隨后,對酶切處理過的DNA片段進行純化與文庫構建,再進行測序,從而獲得全基因組范圍內DNase核酸剪切酶的酶切信息。在酶切信息中,蛋白結合位點處的酶切信息將特異性減弱,就像在DNA上留下一個個足跡一樣,從而可以精確鑒定DNA結合蛋白在DNA分子上的結合位點。
與ChIP-Seq技術相比,DNase-Seq技術的優點非常突出。首先,由于不具有特異性,DNase-Seq可一次性在全基因組范圍內同時檢測多種DNA蛋白的結合位點;其次,由于一次性檢測多種DNA蛋白的結合位點,DNase-Seq大幅提高了檢測效率并降低了檢測成本,使大規模進行DNA蛋白結合位點檢測成為可能;第三,更為重要的是,由于測序起始位置就是酶切位置,DNase-Seq對DNA蛋白結合位點的檢測分辨率可達單堿基。
然而,近期發現DNase核酸剪切酶在切割DNA時存在一定的DNA堿基傾向性,這將對 DNA蛋白結合位點的識別產生不利的影響。如何去除該傾向性已成為基于DNase-Seq的DNA蛋白結合位點識別的一個關鍵問題。
技術實現要素:
本發明的目的在于提供一種濾除DNase高通量測序數據中DNA堿基傾向性偏差的方法。
本發明的目的是這樣實現的:
(1)DNase-Seq實驗數據酶切位點區域DNA堿基獲取
依據DNase-Seq實驗數據在基因組中的位置,提取每一個實驗數據對應酶切位點附近區域的DNA堿基。本發明選用酶切位點附近6個位點的堿基,即以酶切位點為中心,左右各取3個堿基。
(2)DNase-Seq實驗數據DNA堿基傾向性獲取
本發明選用酶切位點附近6個位點的堿基,每個堿基有A、C、G、T等4種取值,則6個位點堿基共有4096種堿基組合。通過統計整個DNase-Seq實驗數據酶切位點處這4096種堿基組合出現的頻次,即可獲得DNase-Seq實驗數據的DNA堿基傾向性。
(3)DNA堿基傾向性去除
設有m個蛋白結合位點,每個結合位點包含n個堿基,則:第i個結合位點的DNase檢測信號為:[Si1,Si2,…,Sin]。其值和為:
考慮DNase的DNA堿基傾向性,則第i個結合位點第j列的DNase檢測信號為:Sij=[(1-w)Pij+wBij]Ri。其中,Pij為第i個結合位點第j列處與DNA結合蛋白的蛋白結構相對應的DNase的固有切割概率,Bij為第i個結合位點第j列處與該處DNA堿基傾向性相對應的DNase的切割概率。Pij是穩定的,可用于DNA蛋白結合位點識別,而Bij是不穩定的,應予以濾除。
具體濾除方法如下:
其中,Sij,Ri可從實驗數據中直接得到。Bij則根據前一步驟獲取的DNase-Seq實驗數據的DNA堿基傾向性得到。w為權值,取值范圍為[0,1]之間,需要進一步確定。
對于m個蛋白結合位點,當權值w取不同值時,會得到不同的[Pi1,Pi2,…,Pin],1≤i≤m。設則當m個[Pi1,Pi2,…,Pin]與[P1,P2,...,Pn]之間的m個相關性值的中位值最大時, 此時的w值為最優值。
本發明的有益效果在于:通過所發明的方法可以精確地濾除DNase高通量測序數據中含有的DNA堿基傾向性偏差,以生成更加準確的DNase-Seq測序結果,從而為后續更高層次的應用分析提供數據保障。
附圖說明
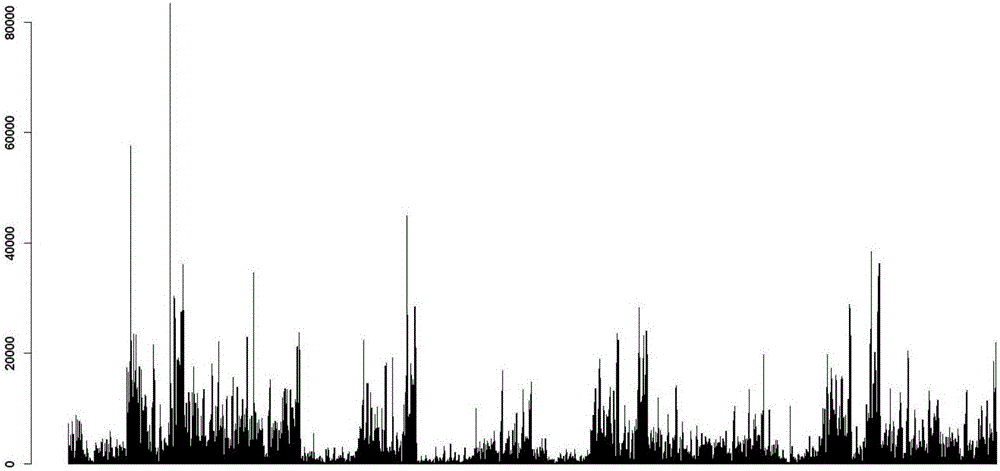
圖1為DNase-Seq實驗數據DNA堿基傾向性直方圖。
圖2為w權值的評價值變化曲線。
圖3為本發明流程圖。
具體實施方式
下面結合附圖對本發明做進一步描述。
作為DNA蛋白結合位點檢測的新技術,DNase-Seq技術具有眾多突出的優點。由于不具有特異性,DNase-Seq可一次性在全基因組范圍內同時檢測多種DNA蛋白的結合位點;由于一次性檢測多種DNA蛋白的結合位點,DNase-Seq大幅提高了檢測效率并降低了檢測成本,使大規模進行DNA蛋白結合位點檢測成為可能;由于測序起始位置就是酶切位置,DNase-Seq對DNA蛋白結合位點的檢測分辨率可達單堿基。
然而,近期發現DNase核酸剪切酶在切割DNA時存在一定的DNA堿基傾向性,這將對DNA蛋白結合位點的識別產生不利的影響。本發明即是針對該問題提出的一種濾除DNase高通量測序數據中DNA堿基傾向性偏差的方法。
1、DNase-Seq實驗數據酶切位點區域DNA堿基獲取
依據DNase-Seq實驗數據在基因組中的位置,提取每一個實驗數據對應酶切位點附近區域的DNA堿基。本發明選用酶切位點附近6個位點的堿基,即以酶切位點為中心,左右各取3個堿基。
2、DNase-Seq實驗數據DNA堿基傾向性獲取
本發明選用酶切位點附近6個位點的堿基,每個堿基有A、C、G、T等4種取值,則6個位點堿基共有4096種堿基組合。通過統計整個DNase-Seq實驗數據酶切位點處這4096種堿基組合出現的頻次,即可獲得DNase-Seq實驗數據的DNA堿基傾向性。
3、DNA堿基傾向性去除
設有m個蛋白結合位點,每個結合位點包含n個堿基,則:第i個結合位點的DNase檢測信號為:[Si1,Si2,…,Sin]。其值和為:
考慮DNase的DNA堿基傾向性,則第i個結合位點第j列的DNase檢測信號為: Sij=[(1-w)Pij+wBij]Ri。其中,Pij為第i個結合位點第j列處與DNA結合蛋白的蛋白結構相對應的DNase的固有切割概率,Bij為第i個結合位點第j列處與該處DNA堿基傾向性相對應的DNase的切割概率。Pij是穩定的,可用于DNA蛋白結合位點識別,而Bij是不穩定的,應予以濾除。
具體濾除方法如下:
其中,Sij,Ri可從實驗數據中直接得到。Bij則根據前一步驟獲取的DNase-Seq實驗數據的DNA堿基傾向性得到。w為權值,取值范圍為[0,1]之間,通過下述方法確定:
對于m個蛋白結合位點,當權值w取不同值時,會得到不同的[Pi1,Pi2,…,Pin],1≤i≤m。設則當m個[Pi1,Pi2,…,Pin]與[P1,P2,...,Pn]之間的m個相關性值的中位值最大時,此時的w值為最優值。
4、實驗驗證
從UCSC國際生物信息網站下載人類基因組堿基序列數據,以及國際ENCODE計劃UW大學測得的人類K562細胞系DNase-Seq測序數據和NFYA轉錄因子ChIP-Seq測序數據。
根據每個DNase-Seq測序數據酶切位點在人類基因組中的位置,提取附近6個位點的堿基,即以酶切位點為中心,左右各取3個堿基。統計酶切位點處4096種堿基組合出現的頻次,獲得DNase-Seq實驗數據的DNA堿基傾向性。該傾向性的直方圖如圖1所示(橫軸為堿基組合,縱軸為頻次)。由圖1可見,DNase-Seq實驗數據存在明顯的DNA堿基傾向性。
根據NFYA轉錄因子的ChIP-Seq測序數據,識別出953個NFYA蛋白結合位點。每個結合位點包含201個堿基。
利用本發明方法對DNase-Seq實驗數據進行DNA堿基傾向性濾除。當w取某一權值時,每個結合位點濾除DNA堿基傾向性的DNase檢測信號為[Pi1,Pi2,…,Pin],1≤i≤953。計算每個結合位點[Pi1,Pi2,…,Pin]與[P1,P2,...,Pn]之間的Pearson相關值,這里n取值為201。選取953個相關值的中位值作為該w值是否優異的評價值。讓w值由0到1變化,獲得如圖2所示的w值的評價值變化曲線(橫軸為w值,縱軸評價值)。由圖2可見,當w值為0.15時,評價值達到最大并不再增加,此時的w值應為最優值,并進而得到與之對應的濾除DNA堿基傾向性的DNase-Seq檢測信息。
作為DNA蛋白結合位點檢測的新技術,DNase-Seq技術具有突出優點。由于不具有特異性,DNase-Seq可一次性在全基因組范圍內同時檢測多種DNA蛋白的結合位點;由于一次性檢測多種DNA蛋白的結合位點,DNase-Seq大幅提高了檢測效率并降低了檢測成本,使大規模進行DNA蛋白結合位點檢測成為可能;由于測序起始位置就是酶切位置,DNase-Seq對DNA蛋白結合位點的檢測分辨率可達單堿基。然而,DNase核酸剪切酶在切割DNA時存在一定的DNA堿基傾向性,這將對DNA蛋白結合位點的識別產生不利的影響。本發明即是針對該問題提出的一種濾除DNase高通量測序數據中DNA堿基傾向性偏差的方法。
- 還沒有人留言評論。精彩留言會獲得點贊!
- 一種構建Hi?C高通量測序文庫的方法與制造工藝
- 桑葚病原菌高通量鑒定及種屬分類方法及其應用與制造工藝
- 基于高通量測序技術檢測乳腺癌/卵巢癌易感基因的多重PCR特異性引物、試劑盒及方法與制造工藝
- 基于高通量測序檢測人EGFR基因變異的質控方法及試劑盒與制造工藝
- 基于高通量測序技術的小麥病原微生物檢測方法及其應用與制造工藝
- 一種基于二代高通量測序技術的地中海貧血癥的基因檢測試劑盒的制造方法與工藝
- 一種基于集群的高通量數據分析方法與制造工藝
- 高通量雙折射干涉成像光譜儀裝置及其成像方法與制造工藝
- 高通量測序檢測人循環腫瘤DNA EGFR基因的捕獲探針及試劑盒的制造方法與工藝
- 一種高通量測序檢測無創親子鑒定的方法與制造工藝