基于高通量測序技術檢測乳腺癌/卵巢癌易感基因的多重PCR特異性引物、試劑盒及方法與流程
本發明屬于基因檢測領域,特別涉及一種基于高通量測序技術檢測乳腺癌/卵巢癌易感基因的多重PCR特異性引物、試劑盒及檢測方法。
背景技術:
乳腺癌是影響婦女健康的最常見惡性腫瘤之一,已經成為女性疾病的第一殺手,發病率逐漸攀升,并逐漸呈現年輕化的趨勢,預計到2030年,我國女性乳腺癌發病率將比2008年上升至31%。同時,乳腺癌有明顯的家族遺傳傾向,基因突變攜帶者的乳腺癌發病風險遠高于一般人群。卵巢癌是致死率最高的婦科惡性腫瘤,發病隱匿,早期診斷困難,除遺傳性卵巢癌外,沒有一級防護措施。
易感基因是指和特定疾病具有一定關聯的基因或等位基因。對乳腺癌/卵巢癌的易感基因及其突變位點進行測序,可得到具體的基因序列信息,進而評估乳腺癌/卵巢癌的遺傳風險,可以做到早預防、早控制,降低疾病發生幾率,提高廣大女性健康水平。大量研究表明,遺傳因素在疾病的易感性中發揮重要作用,復雜疾病的遺傳因素通過微效基因作用的累加可以形成明顯的表型綜合效應。因此對乳腺癌卵巢癌的多個易感基因的遺傳性突變,包括點突變、短片段插入、缺失等進行檢測,并利用生物信息學方法對其進行分析注釋,評估該測試個體是否為高風險人群至關重要。
目前,檢測乳腺癌/卵巢癌易感基因常見方法有Sanger法、基因芯片法、熒光定量PCR法、高通量測序法等。Sanger測序法測序每次只能對一個樣品的某一段區域進行測序,檢測效率低、成本高,且靈敏度低(20%)。基因芯片法、熒光定量PCR法檢測乳腺癌/卵巢癌易感基因,該方法只能針對一個或數個已知突變的位點設計檢測探針形成檢測芯片,因此無法用于檢測未知突變位點,檢測的結果也就不夠全面、準確。高通量測序法主要是由測序儀器提供商提供的測序儀配套文庫構建試劑盒,存在試劑盒貨期長、規格固定、試驗靈活性和準確性不夠等不足。一些試劑廠商根據文獻中分享報道的實驗方法進行建庫,但所完成的實驗效果參差不齊,測序質量不高。
由此可見,現有技術還有待提高。
技術實現要素:
鑒于此,有必要針對上述問題提供一種基于高通量測序技術檢測乳腺癌/卵巢癌易感基因的多重PCR特異性引物、試劑盒和檢測方法,能夠同時檢測多個樣本多個乳腺癌/卵巢癌易感基因,且有效提高檢測效率、準確率,同時降低成本、簡化操作步驟等。
為達到本發明目的,采用以下技術方案:
一種基于高通量測序技術檢測乳腺癌/卵巢癌易感基因的多重PCR特異性引物,包括100對特異性引物,其核苷酸序列如SEQ ID NO:1至SEQ ID NO:200所示。
進一步的,所述特異性引物為經過修飾的引物。
進一步的,所述特異性引物的修飾方法為硫代修飾、脫氧尿嘧啶修飾、5’反向dT修飾中的一種或幾種。
進一步的,所述特異性引物的Tm值為58-62℃。
一種基于高通量測序技術檢測乳腺癌/卵巢癌易感基因的試劑盒,包括上述引物中的至少兩對。
進一步的,所述試劑盒還包括通用引物、酶混合物、質控品、無核酸酶水;所述酶混合物包括:高保真DNA聚合酶、PCRBuffer、dNTP混合物等。
進一步的,所述質控品為人正常基因組DNA。
進一步的,所述特異性引物3’端和5’端分別標記Tag1和Tag2。
進一步的,所述特異性引物3’端Tag1序列與待測樣本目標區域完全互補或部分互補;特異性引物5’端Tag2與通用引物完全互補或部分互補。
進一步的,所述通用引物用于對特異引物擴增目標區域的擴增產物進行再次擴增,對不同待測樣品的目標區域擴增產物進行連接和標記。
進一步的,所述通用引物3’端標記有Tag3;Tag3與特異引物Tag2完全互補或部分互補,長度為50-70bp。
進一步的所述特異性引物或所述試劑盒應用于乳腺癌/卵巢癌易感基因檢測。
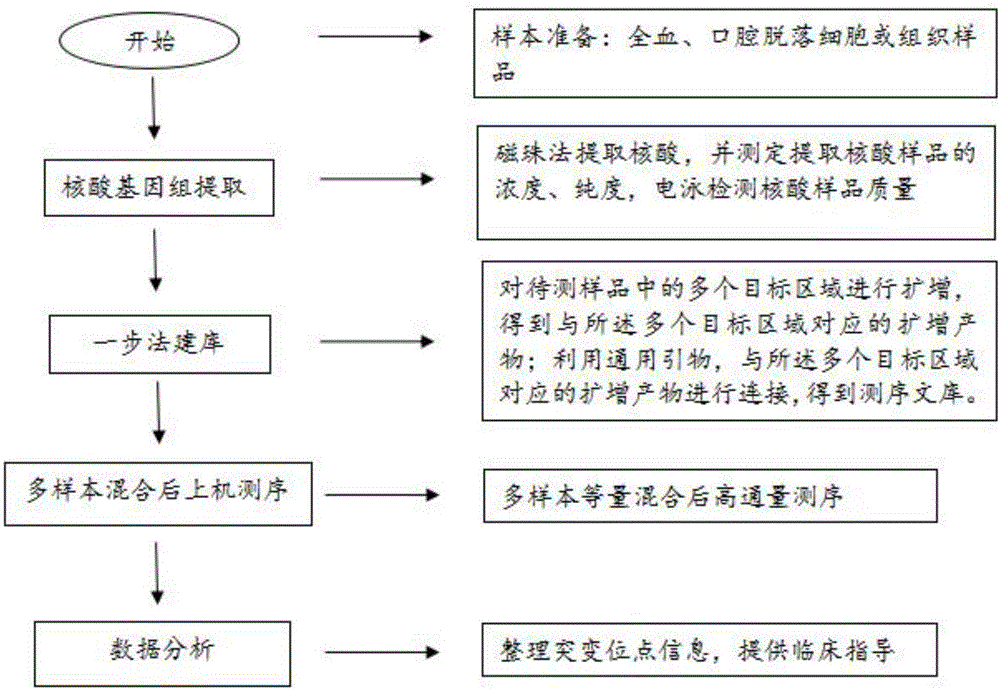
基于高通量測序技術檢測乳腺癌/卵巢癌易感基因的方法,包括以下步驟:
S1:使用磁珠法對樣本進行核酸提取,然后將所提取的樣本核酸進行濃度及純度的測定,測定合格后進行定量稀釋,備用;
S2:用所述特異性引物或所述試劑盒對S1中所提取的核酸的目標區域進行一次PCR擴增,收集擴增產物備用;
S3:使用磁珠法對S2中擴增產物進行核酸純化,進行濃度及純度的測定,然后混樣,定量,上機測序。
進一步的,所述PCR擴增反應體系為:
所述酶混合物包括:高保真DNA聚合酶、PCR Buffer、dNTP混合物等。
進一步的,所述PCR擴增反應程序為:
98℃,激活20min;循環1次;
72℃,延伸10min;循環1次。
進一步的,所述步驟S1中樣本核酸包括乳腺癌/卵巢癌易感基因FGFR2、BRCA1、BRCA2、PALB2、GSTM1、MTHFR、GSTM1或TGF-β1中的至少一種。
進一步的,所述步驟S1中樣本核酸包括乳腺癌/卵巢癌易感基因BRCA1(引物序列SEQ ID NO:1-SEQ ID NO:86)、BRCA2(引物序列SEQ ID NO:87-SEQ ID NO:200)。
進一步的,步驟(2)中所述核酸的目標區域,它們可來源于同一個乳腺癌/卵巢癌易感基因,也可來源于不同的乳腺癌/卵巢癌易感基因;包括但不限于乳腺癌/卵巢癌易感基因上的內部序列、基因的外部調控序列;所述基因內部序列,包括但不限于基因的內含子區域、外顯子區域、同時含有內含子與外顯子的區域。
進一步的,所述檢測方法的檢測樣本包括離體的全血樣品、口腔脫落細胞樣品、組織樣品等;所述組織樣品包括石蠟包埋組織、新鮮組織和冰凍切片等。
上述高通量測序平臺包含但不限于Illumina HiSeq/MiSeq、Life Ion Torrent/Proton平臺、華大基因BGISEQ-500/50等測序平臺。
本發明有益效果:
本發明提供的特異性引物是針對乳腺癌/卵巢癌易感基因的全外顯子設計的引物,結構穩定,覆蓋范圍廣、檢測位點多。每對特異性引物擴增目標區域大小為450-550bp,所有引物Tm值統一設計為58-62℃。
本發明提供的多重PCR引物設計時引進特異性修飾,比如硫代修飾、脫氧尿嘧啶修飾、5’反向dT修飾等修飾方法,修飾后的引物不易被核酸酶降解,確保PCR擴增特異性的同時保證其擴增效率;且具有相對均一的Tm值,在多重擴增時表現出很好的特異性、穩定性及均一性,同時不存在非特異擴增,此方法省去多輪引物篩選的工作。具體的,硫代修飾后的引物序列寡核苷酸鏈上帶雙鍵的氧原子被硫原子取代的衍生物,能夠抵抗核酸酶的降解,增強引物穩定性;脫氧尿嘧啶修飾后的引物寡核苷酸序列中每個脫氧胸腺嘧啶被脫氧尿嘧啶替代,可以使雙鏈溶解溫度增長1.7℃,從而增加雙鏈的穩定性;5’反向dT修飾后的引物寡核苷酸序列5’末端含有反向dT,從而形成交聯,形成的交聯可以抑制在DNA聚合酶延伸過程中的外切核酸酶的降解作用。
本發明自主研發的基于高通量測序技術的針對乳腺癌/卵巢癌發病率較高的易感基因的多重PCR特異性引物、試劑盒和檢測方法,能夠用一次PCR擴增反應捕獲和擴增多個樣本多個乳腺癌/卵巢癌易感基因,包括點突變、短片段插入缺失等遺傳性突變,有效提高檢測效率、準確率,同時降低成本、簡化操作步驟、靈活提供試劑盒包裝規格等。本發明利用高通量基因測序技術突破傳統檢測技術的限制,提高檢測靈敏度;同時還能檢測包括已知突變和未知突變位點的變異情況,得到全面準確的檢測結果。通過對乳腺癌/卵巢癌的易感基因及其突變位點進行測序,可得到具體的基因序列信息,進而評估乳腺癌/卵巢癌的遺傳風險,做到早預防、早控制,降低疾病發生幾率,提高廣大女性健康水平。
本發明采用磁珠純化核酸的方式,省去了傳統建庫中的離心柱式純化,操作簡單;其次,本發明針對高通量測序技術設計的引物只需要一輪PCR,通過選取溫度梯度、時間梯度、循環數經過大量正交實驗,優化PCR反應程序,將樣品傳統建庫的兩次擴增融合在一個PCR反應體系進行,且本發明方法用引物直接在目標基因鏈中捕獲目標區域并進行擴增,不需要對目標基因進行截斷修飾等處理,節省了檢測時間和成本。
附圖說明
圖1檢測乳腺癌/卵巢癌易感基因的方法流程圖。
圖2目標基因建庫PCR擴增的原理圖。
圖3引物特異性驗證結果圖;其中,M:Marker;N:陰性對照;P:陽性對照;1-3:1-3號全血樣品驗證;4-6:4-6號口腔脫落細胞樣品驗證;7-9:7-9號石蠟組織樣品驗證。
圖4引物靈敏度驗證結果圖;其中,M:Marker;N:陰性對照;P:陽性對照;1-3:1號全血樣品三種模板濃度靈敏度驗證;4-6:4號口腔脫落細胞樣品三種模板濃度靈敏度驗證;7-9:7號石蠟組織樣品三種模板濃度靈敏度驗證。
圖5引物重復性驗證結果;其中,M:Marker;N:陰性對照;P:陽性對照;1-3:實驗員A檢測2號全血樣品、5號口腔脫落細胞樣品、8號石蠟組織樣品驗證結果;4-6:實驗員B檢測2號全血樣品、5號口腔脫落細胞樣品、8號石蠟組織樣品驗證結果。
具體實施方式
為了更好地說明本發明所解決的問題、所采用的技術方案和所達到的效果,現結合具體實施例和相關資料進一步闡述。需要說明的是,本發明內容包含但不限于以下實施例及其組合實施方式。
實施例1一種基于高通量測序技術檢測乳腺癌/卵巢癌易感基因的多重PCR特異性引物、試劑盒及方法
1、引物的設計
所述對乳腺癌/卵巢癌易感基因序列選自于University of California Santa Cruz(UCSC)數據庫,選用Primer3引物設計軟件進行易感基因全外顯子引物設計,設計范圍包含了該基因中所有常見和不常見的突變熱點。
本發明對乳腺癌/卵巢癌易感基因全外顯子進行的引物設計共100對,每對特異性引物擴增目標區域大小為450-550bp,覆蓋范圍廣、結構穩定、檢測位點多;特異引物設計時引進特異性修飾,修飾后的引物不易被核酸酶降解,且具有相對均一的Tm值,統一設計為60℃左右(±2℃);所述引物在多重擴增時表現出很好的特異性、穩定性及均一性,同時不存在非特異擴增。具體的,所述引物特異修飾方法為硫代修飾、脫氧尿嘧啶修飾、5’反向dT修飾中的一種或幾種。所述硫代修飾后的引物序列寡核苷酸鏈上帶雙鍵的氧原子被硫原子取代的衍生物,能夠抵抗核酸酶的降解,增強引物穩定性;所述脫氧尿嘧啶修飾后的引物寡核苷酸序列中每個脫氧胸腺嘧啶被脫氧尿嘧啶替代,可以增長雙鏈溶解溫度1.7℃,從而增加雙鏈的穩定性;所述5’反向dT修飾后的引物寡核苷酸序列5’末端含有反向dT,從而形成交聯,形成的交聯可以抑制在DNA聚合酶延伸過程中的外切核酸酶的降解作用。
本發明的測序引物在確保PCR擴增特異性的同時還能保證其擴增效率。
2、檢測乳腺癌/卵巢癌易感基因試劑盒
本發明的檢測乳腺癌/卵巢癌易感基因方法的試劑盒,包括:
特異性引物:用于擴增待測樣品核酸中的多個目標區域,擴增范圍至少覆蓋目標基因的全外顯子區域;
通用引物:用于在文庫構建過程中,對特異引物擴增目標區域的擴增產物進行再次擴增,對不同待測樣品的測序文庫進行標記,進而區分不同樣品;
PCR反應液:包括酶混合物、無核酸酶水;所述酶混合物包括高保真DNA聚合酶(用量為1.25U/反應)、PCR Buffer(用量為1×)、dNTP混合物(用量為200μM/種/反應);
質控品:人正常基因組DNA,來源于正常人全血樣本;本發明質控品來源于市售。
3、基于高通量測序技術檢測乳腺癌/卵巢癌易感基因的方法
包括以下步驟:
S1:利用納米級超順磁性羧基磁珠在不同鹽濃度及疏水環境下與核酸分子可逆吸附的原理,特異吸附核酸分子,通過后續地洗滌及洗脫步驟,獲得高純度的核酸樣本,使用Qubit或NanoDrop進行濃度及純度的測定,根據測定的核酸樣本濃度結果,使用10mM Tris將核酸樣本稀釋至50-250ng/μL,優選的50-100ng/μL作為擴增建庫的起始濃度。
使用1%瓊脂糖凝膠電泳40min,對稀釋后核酸的質量進行評估,根據瓊脂糖凝膠電泳結果的均一性及完整性判斷提取核酸的質量,核酸完整性好或發生輕微降解不影響建庫效果。所提取的核酸濃度、純度測定及1%瓊脂糖凝膠電泳檢測合格后入組并分別進行標記。所述樣本合格的標準為核酸濃度>50ng/μL,OD260/OD280=1.7-2.0,1%瓊脂糖凝膠電泳檢測樣品條帶完整、均一,無明顯拖尾。
S2:用本發明的特異性引物或試劑盒對S1中待測樣品核酸中的易感基因目標區域進行一次PCR擴增,收集擴增產物,擴增產物片段長度為特異引物擴增片段與通用引物之和。
PCR擴增反應體系為:特異引物混合物2μL;通用引物2μL;模板/質控品200ng;酶混合物12.5μL;無核酸酶水將體積補足至25μL。
PCR擴增反應程序為:
98℃,激活20min;循環1次;
72℃,延伸10min;循環1次。
當待測乳腺癌/卵巢癌易感基因有多個時,同一待測樣品的不同乳腺癌/卵巢癌易感基因的擴增可同時進行或部分同時進行。在具體的實驗過程中,可根據需要選用上述任一種方案進行。若分別進行擴增時,需要保證用于構建測序文庫的擴增產物的量是一致的。當待測樣品有多個時,不同樣品的乳腺癌/卵巢癌易感基因的擴增必須分別進行。
S3:利用純化磁珠分別對各樣品的建庫產物進行純化。去除PCR產物中剩余的引物及dNTP等,回收擴增產物;進行濃度、純度測定及1%瓊脂糖凝膠電泳檢測樣品質量,所述樣本合格的標準為核酸濃度>2ng/μL,OD260/OD280=1.7-2.0,1%瓊脂糖凝膠電泳檢測樣品條帶完整、均一,無明顯拖尾。將所有合格核酸樣品(不超過96個)濃度分別稀釋至2nmol/L,并等體積混樣,統一上機測序。
對照試驗:
陽性對照反應體系包括:特異引物混合物2μL;通用引物2μL;酶混合物12.5μL;質控品200ng;無核酸酶水將體積補足至25μL。陽性對照用于檢查反應體系的擴增能力是否正常和樣本是否異常。
陰性對照反應體系包括:特異引物混合物2μL;通用引物2μL;酶混合物12.5μL;無核酸酶水將體積補足至25μL。陰性對照用于檢查反應體系是否存在核酸物質污染。
陽性對照和陰性對照的反應程序同步驟S2的擴增反應程序。
實施例2引物特異性驗證
利用實施例1中S1方法從全血樣品(編號:1-3)、口腔脫落細胞樣品(編號:4-6)、石蠟組織樣品(編號:7-9)中提取核酸,進行濃度、純度測定后,取合格樣品使用10mMTris將每例樣品稀釋至100ng/μL,利用1%瓊脂糖凝膠電泳檢測每例樣品質量(合格標準同實施例1中S1步驟),合格后入組并分別進行標記。利用實施例1中S2方法對上述9例合格樣本進行擴增,上樣量為各2μL,擴增后產物純化后進行1%瓊脂糖凝膠電泳檢測(合格標準同實施例1中S3步驟)。9例樣本使用乳腺癌/卵巢癌易感基因特異引物擴增及檢測方法進行檢測。對照試驗同實施例一,檢測方法與樣本檢測方法相同。檢測結果見圖3。
檢測結果表示,陰性對照除殘留引物外,無任何非特異條帶,說明反應體系是無污染;陽性對照條帶明亮,大小正確,說明反應體系的擴增能力正常。9例樣本最終擴增產物主要集中在600bp,無其他非特異性條帶,與預期設計相符,同時引物二聚體含量極少,且引物二聚體大小與擴增目標片段差異明顯。由此可以看出,本發明的PCR擴增引物及檢測方法能突破傳統方法產生大量引物二聚體的限制,增強了引物特異性。
實施例3引物檢測靈敏度驗證
利用實施例1中S1方法從質檢合格的全血樣品(編號:1)、口腔脫落細胞樣品(編號:4)、石蠟組織樣品(編號:7)提取樣品核酸進行引物檢測靈敏度驗證。每例樣品起始濃度為100ng/μL,按照5倍、10倍、20倍濃度梯度稀釋,稀釋后每例樣品濃度分別為20ng/μL、10ng/μL和5ng/μL,并分別進行樣品名稱及濃度標記。利用實施例1中S2方法對上述9例合格稀釋樣本進行擴增,上樣量為各2μL,9例樣本使用乳腺癌/卵巢癌易感基因特異引物擴增及檢測方法進行檢測。對照試驗同實施例一,檢測方法與樣本檢測方法相同。檢測結果見圖4。
檢測結果表示,陰性對照除殘留引物外,無任何非特異條帶,說明反應體系是無污染;陽性對照條帶明亮,大小正確,說明反應體系的擴增能力正常。9例樣本最終擴增產物主要集中在600bp,無其他非特異性條帶,與預期設計相符,引物二聚體含量極少,且引物二聚體大小與擴增目標片段差異明顯,易分辨。同時,即使模板含量低至10ng,依然能夠成功擴增出目標條帶,由此可以看出,本發明的PCR擴增引物及檢測方法能突破傳統方法的限制,提高了檢測靈敏度。
實施例4引物檢測重復性驗證
利用實施例1中S1方法從質檢合格的全血樣品(編號:2)、口腔脫落細胞樣品(編號:5)、石蠟組織樣品(編號:8)提取樣品核酸進行引物檢測重復性驗證。由2名不同的操作員在2個不同的實驗室平臺按照實施例1中S2方法對上述3例樣品分別進行擴增,上樣量為各2μL,3例樣本使用乳腺癌/卵巢癌易感基因特異引物由不同操作員在不同實驗室得到的6個擴增及檢測方法進行檢測。對照試驗同實施例一,檢測方法與樣本檢測方法相同。檢測結果見圖5。
檢測結果表示,陰性對照除殘留引物外,無任何非特異條帶,說明反應體系是無污染;陽性對照條帶明亮,大小正確,說明反應體系的擴增能力正常。3例樣品共6個最終擴增產物主要集中在600bp,無其他非特異性條帶,與預期設計相符,同時引物二聚體含量極少,且引物二聚體大小與擴增目標片段差異明顯。同時,不同的操作員及不同的實驗平臺檢測結果無明顯差異。由此可以看出,本發明的PCR擴增引物及檢測方法重復性好。
以上所述實施例僅表達了本發明的幾種實施方式,其描述較為具體和詳細,但并不能因此而理解為對本發明專利范圍的限制。應當指出的是,對于本領域的普通技術人員來說,在不脫離本發明構思的前提下,還可以做出若干變形和改進,這些都屬于本發明的保護范圍。因此,本發明專利的保護范圍應以所附權利要求為準。
<110> 廣州奇輝生物科技有限公司
<120> 基于高通量測序技術檢測乳腺癌/卵巢癌易感基因的多重PCR特異性引物、試劑盒及
方法
<160> 200
<170> PatentIn version 3.3
<210> 1
<211> 18
<212> DNA
<213> 人工序列
<400> 1
aatccagagc cccgagag 18
<210> 2
<211> 18
<212> DNA
<213> 人工序列
<400> 2
ccagagcatc acttgggc 18
<210> 3
<211> 27
<212> DNA
<213> 人工序列
<400> 3
ttgtattatt ctaaaacctt ccaaatc 27
<210> 4
<211> 26
<212> DNA
<213> 人工序列
<400> 4
tcatttgcat aggagataat catagg 26
<210> 5
<211> 22
<212> DNA
<213> 人工序列
<400> 5
aaatattgaa cgaacttgag gc 22
<210> 6
<211> 21
<212> DNA
<213> 人工序列
<400> 6
ggtgtttcct gggttatgaa g 21
<210> 7
<211> 20
<212> DNA
<213> 人工序列
<400> 7
tcatggctat ttgccttttg 20
<210> 8
<211> 20
<212> DNA
<213> 人工序列
<400> 8
gtggttgctt ccaacctagc 20
<210> 9
<211> 25
<212> DNA
<213> 人工序列
<400> 9
aaaaggttga taatcacttg ctgag 25
<210> 10
<211> 22
<212> DNA
<213> 人工序列
<400> 10
gagttttcat ggacagcact tg 22
<210> 11
<211> 25
<212> DNA
<213> 人工序列
<400> 11
tgtcttaaca caacaaagag catac 25
<210> 12
<211> 20
<212> DNA
<213> 人工序列
<400> 12
gaggactgct tctagcctgg 20
<210> 13
<211> 19
<212> DNA
<213> 人工序列
<400> 13
gccaacaatt gcttgactg 19
<210> 14
<211> 26
<212> DNA
<213> 人工序列
<400> 14
ttggcaaaac tataagataa ggaatc 26
<210> 15
<211> 21
<212> DNA
<213> 人工序列
<400> 15
ctagcattgt acctgccaca g 21
<210> 16
<211> 20
<212> DNA
<213> 人工序列
<400> 16
tgcacataca tccctgaacc 20
<210> 17
<211> 20
<212> DNA
<213> 人工序列
<400> 17
tggtcatttg acagttctgc 20
<210> 18
<211> 25
<212> DNA
<213> 人工序列
<400> 18
ctgtatctac ccactctctt ttcag 25
<210> 19
<211> 20
<212> DNA
<213> 人工序列
<400> 19
gttgatttcc acctccaagg 20
<210> 20
<211> 20
<212> DNA
<213> 人工序列
<400> 20
tgtttcctta cttccagccc 20
<210> 21
<211> 20
<212> DNA
<213> 人工序列
<400> 21
gcacaaatac tcatgccagc 20
<210> 22
<211> 22
<212> DNA
<213> 人工序列
<400> 22
cagctacttt ggcatttgat tc 22
<210> 23
<211> 25
<212> DNA
<213> 人工序列
<400> 23
tgttccttgg ataacactaa atagc 25
<210> 24
<211> 21
<212> DNA
<213> 人工序列
<400> 24
tggctcagta acaaatgctc c 21
<210> 25
<211> 23
<212> DNA
<213> 人工序列
<400> 25
tgaaagagtt cactccaaat cag 23
<210> 26
<211> 20
<212> DNA
<213> 人工序列
<400> 26
agctttcgtt ttgaaagcag 20
<210> 27
<211> 20
<212> DNA
<213> 人工序列
<400> 27
caagaaagca gatttggcag 20
<210> 28
<211> 18
<212> DNA
<213> 人工序列
<400> 28
tgcctgactg gcatttgg 18
<210> 29
<211> 21
<212> DNA
<213> 人工序列
<400> 29
ggaggaagtc ttctaccagg c 21
<210> 30
<211> 22
<212> DNA
<213> 人工序列
<400> 30
ttctcttgga aggctaggat tg 22
<210> 31
<211> 20
<212> DNA
<213> 人工序列
<400> 31
cagcgatact ttcccagagc 20
<210> 32
<211> 20
<212> DNA
<213> 人工序列
<400> 32
cttggggttt tcaaatgctg 20
<210> 33
<211> 22
<212> DNA
<213> 人工序列
<400> 33
gcagtatttc attggtacct gg 22
<210> 34
<211> 18
<212> DNA
<213> 人工序列
<400> 34
gacccagagt gggcagag 18
<210> 35
<211> 20
<212> DNA
<213> 人工序列
<400> 35
acattcaagg tttcaaagcg 20
<210> 36
<211> 26
<212> DNA
<213> 人工序列
<400> 36
ttgtaaaagt ccatgtttat ttggag 26
<210> 37
<211> 23
<212> DNA
<213> 人工序列
<400> 37
ccagttgata atgccaaatg tag 23
<210> 38
<211> 22
<212> DNA
<213> 人工序列
<400> 38
gagcccactt cattagtact gg 22
<210> 39
<211> 20
<212> DNA
<213> 人工序列
<400> 39
acagtgagca caattagccg 20
<210> 40
<211> 21
<212> DNA
<213> 人工序列
<400> 40
gcatgactac ttcccatagg c 21
<210> 41
<211> 23
<212> DNA
<213> 人工序列
<400> 41
cctggaagta attgtaagca tcc 23
<210> 42
<211> 20
<212> DNA
<213> 人工序列
<400> 42
aacaagtgtt ggaagcaggg 20
<210> 43
<211> 19
<212> DNA
<213> 人工序列
<400> 43
gcaaaagcgt ccagaaagg 19
<210> 44
<211> 22
<212> DNA
<213> 人工序列
<400> 44
tgtgaagaaa acaagctagc ag 22
<210> 45
<211> 23
<212> DNA
<213> 人工序列
<400> 45
cttaaatgac tgcagtaacc agg 23
<210> 46
<211> 24
<212> DNA
<213> 人工序列
<400> 46
tcacttctat aaatagactg gggc 24
<210> 47
<211> 22
<212> DNA
<213> 人工序列
<400> 47
cagcaagttg cagcgtttat ag 22
<210> 48
<211> 20
<212> DNA
<213> 人工序列
<400> 48
gcaaaggaca ccacacacac 20
<210> 49
<211> 27
<212> DNA
<213> 人工序列
<400> 49
acttgtagtt ccatactagg tgatttc 27
<210> 50
<211> 20
<212> DNA
<213> 人工序列
<400> 50
ttacccatgt gctgagcaag 20
<210> 51
<211> 26
<212> DNA
<213> 人工序列
<400> 51
ttgtgtatca tagattgatg cttttg 26
<210> 52
<211> 24
<212> DNA
<213> 人工序列
<400> 52
gcaataaaag tgtataaatg cctg 24
<210> 53
<211> 20
<212> DNA
<213> 人工序列
<400> 53
gtcctttcac aattggtggc 20
<210> 54
<211> 26
<212> DNA
<213> 人工序列
<400> 54
cagagtaaaa tcaaagtgtt tgttcc 26
<210> 55
<211> 24
<212> DNA
<213> 人工序列
<400> 55
tgtaattcaa cattcatcgt tgtg 24
<210> 56
<211> 20
<212> DNA
<213> 人工序列
<400> 56
tccattgcat tatacccagc 20
<210> 57
<211> 20
<212> DNA
<213> 人工序列
<400> 57
ctgaagacag agccccagag 20
<210> 58
<211> 27
<212> DNA
<213> 人工序列
<400> 58
tgacaatacc tacataaaac tctttcc 27
<210> 59
<211> 24
<212> DNA
<213> 人工序列
<400> 59
tctgagctgt gtgctagagg taac 24
<210> 60
<211> 20
<212> DNA
<213> 人工序列
<400> 60
tttatgcagc agatgcaagg 20
<210> 61
<211> 23
<212> DNA
<213> 人工序列
<400> 61
cacttcctga ttttgttttc aac 23
<210> 62
<211> 20
<212> DNA
<213> 人工序列
<400> 62
aggtgttaaa gggaggaggg 20
<210> 63
<211> 20
<212> DNA
<213> 人工序列
<400> 63
atctccgtga aaagagcacg 20
<210> 64
<211> 26
<212> DNA
<213> 人工序列
<400> 64
atgactgaat gaatatctct ggttag 26
<210> 65
<211> 22
<212> DNA
<213> 人工序列
<400> 65
ctctttctct tatcctgatg gg 22
<210> 66
<211> 20
<212> DNA
<213> 人工序列
<400> 66
tgcaaagggg agtggaatac 20
<210> 67
<211> 20
<212> DNA
<213> 人工序列
<400> 67
tttccttctc tccattcccc 20
<210> 68
<211> 20
<212> DNA
<213> 人工序列
<400> 68
taagacaaag gctggtgctg 20
<210> 69
<211> 20
<212> DNA
<213> 人工序列
<400> 69
ggtagagggc ctgggttaag 20
<210> 70
<211> 23
<212> DNA
<213> 人工序列
<400> 70
gaatattgtg tcctccctct ctg 23
<210> 71
<211> 23
<212> DNA
<213> 人工序列
<400> 71
tcctactttg acactttgaa tgc 23
<210> 72
<211> 19
<212> DNA
<213> 人工序列
<400> 72
aaacccatgc aaaaggacc 19
<210> 73
<211> 21
<212> DNA
<213> 人工序列
<400> 73
tccaggagaa tgaattgaca c 21
<210> 74
<211> 20
<212> DNA
<213> 人工序列
<400> 74
agccagaagt ccttttcagg 20
<210> 75
<211> 21
<212> DNA
<213> 人工序列
<400> 75
ggaccccaag aatgagctta c 21
<210> 76
<211> 20
<212> DNA
<213> 人工序列
<400> 76
tctgtgcttc cagccctaag 20
<210> 77
<211> 20
<212> DNA
<213> 人工序列
<400> 77
ttaaacgcca ccaattgagc 20
<210> 78
<211> 20
<212> DNA
<213> 人工序列
<400> 78
atgcccaggt ttcaagtttc 20
<210> 79
<211> 20
<212> DNA
<213> 人工序列
<400> 79
tgcttgggat cgattatgtg 20
<210> 80
<211> 18
<212> DNA
<213> 人工序列
<400> 80
cccgggctga agtgattc 18
<210> 81
<211> 19
<212> DNA
<213> 人工序列
<400> 81
caaggtgggc agatcactg 19
<210> 82
<211> 27
<212> DNA
<213> 人工序列
<400> 82
gaatggatta tataccagag ctacaac 27
<210> 83
<211> 20
<212> DNA
<213> 人工序列
<400> 83
agcctgggtg acagtgagac 20
<210> 84
<211> 23
<212> DNA
<213> 人工序列
<400> 84
ccaacagcta taaacagtcc tgg 23
<210> 85
<211> 20
<212> DNA
<213> 人工序列
<400> 85
caccaggaag gaagctgttg 20
<210> 86
<211> 20
<212> DNA
<213> 人工序列
<400> 86
aaagtcggct ggcctaagtc 20
<210> 87
<211> 17
<212> DNA
<213> 人工序列
<400> 87
ctgacttccg ggtggtg 17
<210> 88
<211> 19
<212> DNA
<213> 人工序列
<400> 88
gactgggact gcggaagac 19
<210> 89
<211> 20
<212> DNA
<213> 人工序列
<400> 89
caaattttcc agcgcttctg 20
<210> 90
<211> 21
<212> DNA
<213> 人工序列
<400> 90
agcaacactg tgacgtactg g 21
<210> 91
<211> 25
<212> DNA
<213> 人工序列
<400> 91
tgtctgtcac tggttaaaac taagg 25
<210> 92
<211> 22
<212> DNA
<213> 人工序列
<400> 92
aacacaggtt tgcctaaatt cc 22
<210> 93
<211> 25
<212> DNA
<213> 人工序列
<400> 93
ttctcattcc cagtatagag gagac 25
<210> 94
<211> 24
<212> DNA
<213> 人工序列
<400> 94
aagatcttct accaggctct tagc 24
<210> 95
<211> 21
<212> DNA
<213> 人工序列
<400> 95
ccagcagctg aaatttgtga g 21
<210> 96
<211> 21
<212> DNA
<213> 人工序列
<400> 96
tctcagggca aaggtataac g 21
<210> 97
<211> 22
<212> DNA
<213> 人工序列
<400> 97
tccttaatga tcagggcatt tc 22
<210> 98
<211> 23
<212> DNA
<213> 人工序列
<400> 98
tgacaattat caacctcatc tgc 23
<210> 99
<211> 23
<212> DNA
<213> 人工序列
<400> 99
ttcactgtgt tgattgacct ttc 23
<210> 100
<211> 22
<212> DNA
<213> 人工序列
<400> 100
gacagcagag tttcacagga ag 22
<210> 101
<211> 20
<212> DNA
<213> 人工序列
<400> 101
tggacctagg ttgattgcag 20
<210> 102
<211> 20
<212> DNA
<213> 人工序列
<400> 102
aggcagaggt tgcggtaaac 20
<210> 103
<211> 20
<212> DNA
<213> 人工序列
<400> 103
caggagaagg ggtgactgac 20
<210> 104
<211> 20
<212> DNA
<213> 人工序列
<400> 104
accattcaca ggccaaagac 20
<210> 105
<211> 20
<212> DNA
<213> 人工序列
<400> 105
tggaaagtca atgccaaatg 20
<210> 106
<211> 21
<212> DNA
<213> 人工序列
<400> 106
ggctagaaat acgtggcaaa g 21
<210> 107
<211> 20
<212> DNA
<213> 人工序列
<400> 107
gttgtaccgt ctttggcctg 20
<210> 108
<211> 23
<212> DNA
<213> 人工序列
<400> 108
cttgcattga aagtctcttt agg 23
<210> 109
<211> 21
<212> DNA
<213> 人工序列
<400> 109
tgaagagcag catcttgaat c 21
<210> 110
<211> 20
<212> DNA
<213> 人工序列
<400> 110
aaacctgcat tcttcaaagc 20
<210> 111
<211> 20
<212> DNA
<213> 人工序列
<400> 111
tgtttgctca cagaaggagg 20
<210> 112
<211> 25
<212> DNA
<213> 人工序列
<400> 112
cagaaggaat cgtcatctat aaaac 25
<210> 113
<211> 26
<212> DNA
<213> 人工序列
<400> 113
gtgattgatg gtactttaat tttgtc 26
<210> 114
<211> 19
<212> DNA
<213> 人工序列
<400> 114
atgctgcagc caagacctc 19
<210> 115
<211> 25
<212> DNA
<213> 人工序列
<400> 115
cagttattta ttaccccaga agctg 25
<210> 116
<211> 24
<212> DNA
<213> 人工序列
<400> 116
gcacatacat cttgattctt ttcc 24
<210> 117
<211> 22
<212> DNA
<213> 人工序列
<400> 117
tcctacttcc aaggatgttc tg 22
<210> 118
<211> 25
<212> DNA
<213> 人工序列
<400> 118
aagcaagatt attcctttca ttagc 25
<210> 119
<211> 22
<212> DNA
<213> 人工序列
<400> 119
caattcaacc aaaacacaaa tc 22
<210> 120
<211> 20
<212> DNA
<213> 人工序列
<400> 120
ttggacctaa gagtcctgcc 20
<210> 121
<211> 22
<212> DNA
<213> 人工序列
<400> 121
ttggtttatg ttcttgcaga gg 22
<210> 122
<211> 21
<212> DNA
<213> 人工序列
<400> 122
aggcttgctc agtttctttt g 21
<210> 123
<211> 26
<212> DNA
<213> 人工序列
<400> 123
aacataacat taagaagagc aaaatg 26
<210> 124
<211> 21
<212> DNA
<213> 人工序列
<400> 124
catctggttt tcaggcactt c 21
<210> 125
<211> 20
<212> DNA
<213> 人工序列
<400> 125
aacacctagc caaaaggcag 20
<210> 126
<211> 24
<212> DNA
<213> 人工序列
<400> 126
tgcagagctt cagtagaaac attc 24
<210> 127
<211> 20
<212> DNA
<213> 人工序列
<400> 127
ttaaacggaa gtttgctggc 20
<210> 128
<211> 24
<212> DNA
<213> 人工序列
<400> 128
tttctactgg cagcagtata tttg 24
<210> 129
<211> 26
<212> DNA
<213> 人工序列
<400> 129
ttcaagtaaa tgtcatgatt ctgttg 26
<210> 130
<211> 20
<212> DNA
<213> 人工序列
<400> 130
cttgagcttt cgcaacttcc 20
<210> 131
<211> 23
<212> DNA
<213> 人工序列
<400> 131
ttcataaaga tgaaacggac ttg 23
<210> 132
<211> 25
<212> DNA
<213> 人工序列
<400> 132
tgtttcctca taacttagaa tgtcc 25
<210> 133
<211> 25
<212> DNA
<213> 人工序列
<400> 133
aaaatattag tgtcgccaaa gagtc 25
<210> 134
<211> 20
<212> DNA
<213> 人工序列
<400> 134
tttagggtct ttgcccattg 20
<210> 135
<211> 20
<212> DNA
<213> 人工序列
<400> 135
aacccgaacg tgatgaaaag 20
<210> 136
<211> 24
<212> DNA
<213> 人工序列
<400> 136
gatttgtgta acaagttgca ggac 24
<210> 137
<211> 20
<212> DNA
<213> 人工序列
<400> 137
agctgcccca aagtgtaaag 20
<210> 138
<211> 25
<212> DNA
<213> 人工序列
<400> 138
tcagaatggt aggaatagct gttag 25
<210> 139
<211> 22
<212> DNA
<213> 人工序列
<400> 139
ttcagtcatt gaaaattcag cc 22
<210> 140
<211> 21
<212> DNA
<213> 人工序列
<400> 140
cagtttgtgg gtatgcattt g 21
<210> 141
<211> 22
<212> DNA
<213> 人工序列
<400> 141
cttgattctg gtattgagcc ag 22
<210> 142
<211> 20
<212> DNA
<213> 人工序列
<400> 142
aatgcctcgt aacaacctgc 20
<210> 143
<211> 23
<212> DNA
<213> 人工序列
<400> 143
tggtaaaatc gtttgtgttt cac 23
<210> 144
<211> 26
<212> DNA
<213> 人工序列
<400> 144
gactgactta tgaagcttcc ctatac 26
<210> 145
<211> 20
<212> DNA
<213> 人工序列
<400> 145
tgaatgtagc acgcattcac 20
<210> 146
<211> 21
<212> DNA
<213> 人工序列
<400> 146
aaacttgctt tccacttgct g 21
<210> 147
<211> 26
<212> DNA
<213> 人工序列
<400> 147
aagatagtac caagcaagtc ttttcc 26
<210> 148
<211> 23
<212> DNA
<213> 人工序列
<400> 148
ttttctgaag aaccaccttc aac 23
<210> 149
<211> 24
<212> DNA
<213> 人工序列
<400> 149
ttaagggagt gttagaggaa tttg 24
<210> 150
<211> 23
<212> DNA
<213> 人工序列
<400> 150
gcaatttcta ctgcttctgt ttc 23
<210> 151
<211> 20
<212> DNA
<213> 人工序列
<400> 151
ttgggaaaag aacaggcttc 20
<210> 152
<211> 24
<212> DNA
<213> 人工序列
<400> 152
tctgtatatc aaaccatact cccc 24
<210> 153
<211> 26
<212> DNA
<213> 人工序列
<400> 153
tgaagactga ctttactctt tcaaac 26
<210> 154
<211> 20
<212> DNA
<213> 人工序列
<400> 154
taggcacagt ggctcatgtc 20
<210> 155
<211> 23
<212> DNA
<213> 人工序列
<400> 155
tgctgatttc tgttgtatgc ttg 23
<210> 156
<211> 24
<212> DNA
<213> 人工序列
<400> 156
caatttcctt aattcctctc aacc 24
<210> 157
<211> 18
<212> DNA
<213> 人工序列
<400> 157
agggtctgca acaaaggc 18
<210> 158
<211> 22
<212> DNA
<213> 人工序列
<400> 158
cttttgtctg ttttcctcca ag 22
<210> 159
<211> 24
<212> DNA
<213> 人工序列
<400> 159
tttcaggaca tccattttat caag 24
<210> 160
<211> 24
<212> DNA
<213> 人工序列
<400> 160
catcaggaca ttatttaaca acgg 24
<210> 161
<211> 18
<212> DNA
<213> 人工序列
<400> 161
gtgagccact gtgcctgg 18
<210> 162
<211> 22
<212> DNA
<213> 人工序列
<400> 162
ccattcctgc actaatgtgt tc 22
<210> 163
<211> 24
<212> DNA
<213> 人工序列
<400> 163
tttggtaaat tcagttttgg tttg 24
<210> 164
<211> 21
<212> DNA
<213> 人工序列
<400> 164
gagaagaaag agggatgagg g 21
<210> 165
<211> 19
<212> DNA
<213> 人工序列
<400> 165
accatgctca gcaatgaag 19
<210> 166
<211> 20
<212> DNA
<213> 人工序列
<400> 166
cactgacaac tggcttgtgc 20
<210> 167
<211> 27
<212> DNA
<213> 人工序列
<400> 167
tctagagtca cacttcctaa aatatgc 27
<210> 168
<211> 20
<212> DNA
<213> 人工序列
<400> 168
gaggggagga tctaactggg 20
<210> 169
<211> 26
<212> DNA
<213> 人工序列
<400> 169
gcgcaaatat atctgaaact tctagc 26
<210> 170
<211> 27
<212> DNA
<213> 人工序列
<400> 170
tctagaattt aactgaatca atgactg 27
<210> 171
<211> 25
<212> DNA
<213> 人工序列
<400> 171
ggcagttcta gaagaatgaa aactc 25
<210> 172
<211> 19
<212> DNA
<213> 人工序列
<400> 172
ggcaagagac cgaaactcc 19
<210> 173
<211> 20
<212> DNA
<213> 人工序列
<400> 173
tccactaatc tcagcctccc 20
<210> 174
<211> 22
<212> DNA
<213> 人工序列
<400> 174
tgtcccttgt tgctattctt tg 22
<210> 175
<211> 20
<212> DNA
<213> 人工序列
<400> 175
tttgggtgtt ttatgcttgg 20
<210> 176
<211> 20
<212> DNA
<213> 人工序列
<400> 176
cctgtgatgg ccagagagtc 20
<210> 177
<211> 26
<212> DNA
<213> 人工序列
<400> 177
tgtcctgttt aaagccatct agttac 26
<210> 178
<211> 25
<212> DNA
<213> 人工序列
<400> 178
tggattttgc ttctctgata taaac 25
<210> 179
<211> 24
<212> DNA
<213> 人工序列
<400> 179
gataatcact tcttccattg catc 24
<210> 180
<211> 24
<212> DNA
<213> 人工序列
<400> 180
tggagattcc ataaactaac aagc 24
<210> 181
<211> 27
<212> DNA
<213> 人工序列
<400> 181
gataagtgct tgttagttta tggaatc 27
<210> 182
<211> 25
<212> DNA
<213> 人工序列
<400> 182
tggtagctcc aactaatcat aagag 25
<210> 183
<211> 25
<212> DNA
<213> 人工序列
<400> 183
ggcatattag agtttccttt cttgc 25
<210> 184
<211> 25
<212> DNA
<213> 人工序列
<400> 184
aagctatttc cttgatactg gactg 25
<210> 185
<211> 22
<212> DNA
<213> 人工序列
<400> 185
ggcatgtttg acaattggta tc 22
<210> 186
<211> 22
<212> DNA
<213> 人工序列
<400> 186
aacagaatat acgatggcct cc 22
<210> 187
<211> 20
<212> DNA
<213> 人工序列
<400> 187
taggggaggg agactgtgtg 20
<210> 188
<211> 20
<212> DNA
<213> 人工序列
<400> 188
actccttggt ggctgaaatg 20
<210> 189
<211> 22
<212> DNA
<213> 人工序列
<400> 189
gaagagcctt ggatttcttg ag 22
<210> 190
<211> 20
<212> DNA
<213> 人工序列
<400> 190
tcttctgaac tggtgggagc 20
<210> 191
<211> 21
<212> DNA
<213> 人工序列
<400> 191
gctgacgaag aacttgcatt g 21
<210> 192
<211> 20
<212> DNA
<213> 人工序列
<400> 192
ccgatacaca aacgctgagg 20
<210> 193
<211> 25
<212> DNA
<213> 人工序列
<400> 193
aaaataagca ggacacaatt acaac 25
<210> 194
<211> 20
<212> DNA
<213> 人工序列
<400> 194
actcctccca cctcagcttc 20
<210> 195
<211> 18
<212> DNA
<213> 人工序列
<400> 195
ttgcccgatt ccgtattg 18
<210> 196
<211> 27
<212> DNA
<213> 人工序列
<400> 196
gctaaactaa aaggaattat ctgcatc 27
<210> 197
<211> 19
<212> DNA
<213> 人工序列
<400> 197
ggcaacatag ggagacccc 19
<210> 198
<211> 25
<212> DNA
<213> 人工序列
<400> 198
aaaggaatta gagttacact gaggg 25
<210> 199
<211> 22
<212> DNA
<213> 人工序列
<400> 199
ttccatcctg ttcaaaagtc ag 22
<210> 200
<211> 20
<212> DNA
<213> 人工序列
<400> 200
gctctgggtc ctgtgatagc 20
- 還沒有人留言評論。精彩留言會獲得點贊!
- 一種構建Hi?C高通量測序文庫的方法與制造工藝
- 桑葚病原菌高通量鑒定及種屬分類方法及其應用與制造工藝
- 基于高通量測序技術檢測乳腺癌/卵巢癌易感基因的多重PCR特異性引物、試劑盒及方法與制造工藝
- 基于高通量測序檢測人EGFR基因變異的質控方法及試劑盒與制造工藝
- 基于高通量測序技術的小麥病原微生物檢測方法及其應用與制造工藝
- 一種基于二代高通量測序技術的地中海貧血癥的基因檢測試劑盒的制造方法與工藝
- 一種基于集群的高通量數據分析方法與制造工藝
- 高通量雙折射干涉成像光譜儀裝置及其成像方法與制造工藝
- 高通量測序檢測人循環腫瘤DNA EGFR基因的捕獲探針及試劑盒的制造方法與工藝
- 一種高通量測序檢測無創親子鑒定的方法與制造工藝